应用机器学习的快速掌握之道是练习端到端的项目。
在本贴中,您将学习如何在 Weka 中端到端地完成一个二元分类问题。阅读本贴后,您将了解
- 如何加载数据集并分析加载的数据。
- 如何创建数据的多个不同变换视图,并在每个视图上评估一系列算法。
- 如何最终确定模型并呈现结果,以便对新数据进行预测。
通过我的新书 《Weka 机器学习精通》 来启动您的项目,其中包含一步一步的教程和所有示例的清晰截图。
让我们开始吧。

Weka 二元分类分步教程
照片由 Anita Ritenour 提供,部分权利保留。
教程概述
本教程将引导您完成完成机器学习项目所需的关键步骤。
我们将按照以下流程进行
- 加载数据集。
- 分析数据集。
- 准备数据集的视图。
- 评估算法。
- 最终确定模型并呈现结果。
在Weka机器学习方面需要更多帮助吗?
参加我为期14天的免费电子邮件课程,逐步探索如何使用该平台。
点击注册,同时获得该课程的免费PDF电子书版本。
1. 加载数据集
本教程中使用的问题是 Pima 印第安人糖尿病发病数据集。
在此数据集中,每个实例代表一名患者的医疗详细信息,任务是预测该患者在未来五年内是否会患上糖尿病。有 8 个数值输入变量,所有变量的尺度各不相同。
最佳结果的准确率约为 77%。
1. 打开 Weka GUI Chooser。
2. 单击“Explorer”按钮打开 Weka Explorer。
3. 单击“Open file…”按钮,导航到 data/ 目录并选择 diabetes.arff。单击“Open”按钮。
数据集现已加载到 Weka 中。
Weka 加载 Pima 印第安人糖尿病发病数据集
2. 分析数据集
在开始建模之前,检查数据非常重要。
检查每个属性的分布以及属性之间的交互作用,可能会为我们可能使用的数据转换和建模技术提供线索。
描述性统计
在“Current relation”窗格中查看数据集的详细信息。我们可以注意到几点:
- 数据集的名称为 pima_diabetes。
- 数据集中有 768 个实例。如果我们使用 10 折交叉验证来评估模型,那么每一折大约有 76 个实例,这是可以接受的。
- 有 9 个属性,8 个输入属性和一个输出属性。
点击“Attributes”窗格中的每个属性,并查看“Selected attribute”窗格中的摘要统计信息。
我们可以注意到我们数据的一些事实:
- 输入属性均为数值型,且尺度不同。对数据进行归一化或标准化可能会带来一些好处。
- 没有标记的缺失值。
- 某些属性的值似乎不合理,特别是:plas、pres、skin、insu 和 mass 的值为 0。这些很可能是标记有误的缺失数据。
- 类别属性是标称型,有两个输出值,这意味着这是一个二类或二元分类问题。
- 类别属性不平衡,1 个“阳性”结果对应 1.8 个“阴性”结果,阴性病例数量接近阳性病例的两倍。平衡类别值可能会有所帮助。
属性分布
点击“Visualize All”按钮,让我们来查看每个属性的图形分布。
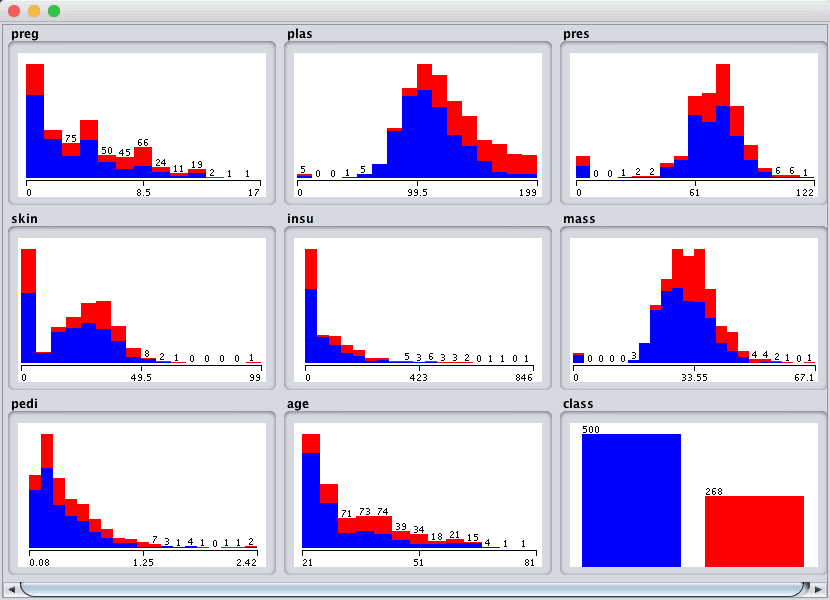
Weka Pima 印第安人单变量属性分布
我们可以注意到数据形状的一些特点:
- 某些属性具有类似高斯分布的特征,例如 plas、pres、skin 和 mass,这表明具有此类假设的方法(如逻辑回归和朴素贝叶斯)可能会取得良好的结果。
- 我们看到不同属性值上的类之间有很大的重叠。类似乎不易分离。
- 我们可以通过图形清晰地看到类的不平衡。
属性交互
点击“Visualize”选项卡,让我们来查看属性之间的一些交互。
- 增加窗口大小以显示所有图。
- 将“PointSize”增加到 3,使点更容易看到。
- 点击“Update”按钮应用更改。
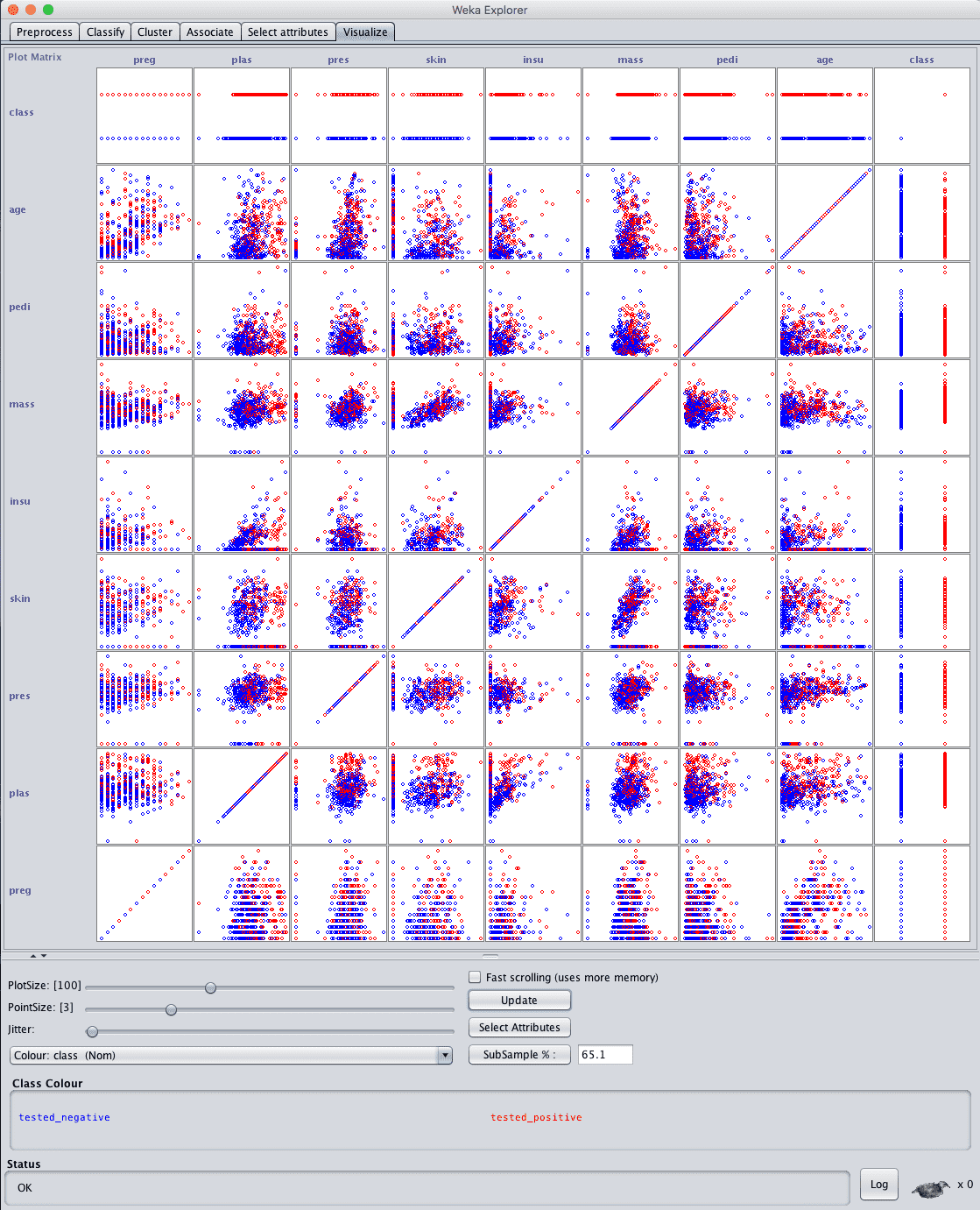
Weka Pima 印第安人散点图矩阵
查看输入变量的图,我们通常可以看到散点图上类之间分离性较差。这个数据集并非易事。
这表明进行良好的数据转换和创建数据集的多个视图可能对我们有益。这也表明使用集成方法可能会带来好处。
3. 准备数据集的视图
我们在上一节中注意到,这可能是一个困难的问题,并且多个数据视图可能对我们有益。
在本节中,我们将创建数据的不同视图,以便在下一节评估算法时,我们可以了解哪些视图在将分类问题的结构暴露给模型方面通常更好。
我们将创建 3 个额外的数据视图,因此除了原始数据之外,我们总共有 4 个数据集副本。我们将从原始数据创建每个数据集视图,并将其保存到新文件中,以便在我们的实验中稍后使用。
归一化视图
我们将创建的第一个视图是所有输入属性均归一化到 0 到 1 的范围。这可能对受属性尺度影响的多种算法(如回归和基于实例的方法)有益。
- 在 Explorer 中,加载 data/diabetes.arff 文件。
- 在“Filter”窗格中单击“Choose”按钮,然后选择“unsupervised.attribute.Normalize”过滤器。
- 单击“Apply”按钮应用过滤器。
- 单击“Attributes”窗格中的每个属性,并在“Selected attribute”窗格中查看最小值和最大值,以确认它们是 0 和 1。
- 单击“Save…”按钮,导航到合适的目录,然后输入此转换后数据集的合适名称,例如“diabetes-normalize.arff”。
- 关闭 Explorer 界面,以避免污染我们想要创建的其他视图。
Weka 归一化 Pima 印第安人数据集
标准化视图
我们在上一节中注意到,有些属性具有类似高斯分布的特征。我们可以通过使用标准化过滤器来重新缩放数据并考虑此分布。
这将创建一个数据集副本,其中每个属性的平均值为 0,标准差(平均方差)为 1。这可能对下一节中假设输入属性具有高斯分布的算法(如逻辑回归和朴素贝叶斯)有益。
- 打开Weka Explorer。
- 加载 Pima 印第安人糖尿病发病数据集。
- 在“Filter”窗格中单击“Choose”按钮,然后选择“unsupervised.attribute.Standardize”过滤器。
- 单击“Apply”按钮应用过滤器。
- 单击“Attributes”窗格中的每个属性,并在“Selected attribute”窗格中查看平均值和标准差,以确认它们分别是 0 和 1。
- 单击“Save…”按钮,导航到合适的目录,然后输入此转换后数据集的合适名称,例如“diabetes-standardize.arff”。
- 关闭 Explorer 界面。
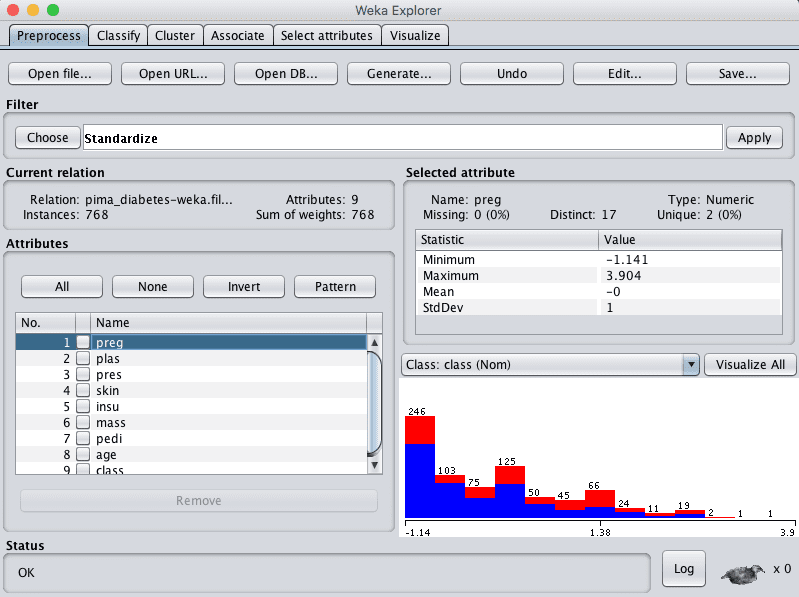
Weka 标准化 Pima 印第安人数据集
缺失数据
在前一节中,我们怀疑某些属性中的 0 值被视为无效或缺失数据。
我们可以创建一个新的数据集副本,标记缺失数据,然后用每个属性的平均值进行插补。这可能有助于那些假设属性分布平滑变化的算法,例如逻辑回归和基于实例的方法。
首先,让我们将一些属性的 0 值标记为缺失。
- 打开Weka Explorer。
- 加载 Pima 印第安人糖尿病发病数据集。
- 单击 Filter 的“Choose”按钮,然后选择 unsupervised.attribute.NumericalCleaner 过滤器。
- 单击过滤器以配置它。
- 将 attributeIndices 设置为 2-6
- 将 minThreshold 设置为 0.1E-8(接近零),这是每个属性允许的最小值。
- 将 minDefault 设置为 NaN,表示未知,它将替换低于阈值的值。
- 单击过滤器配置上的“OK”按钮。
- 单击“Apply”按钮应用过滤器。
- 单击“Attributes”窗格中的每个属性,并查看每个属性的缺失值数量。您应该会看到属性 2 到 6 存在非零计数。
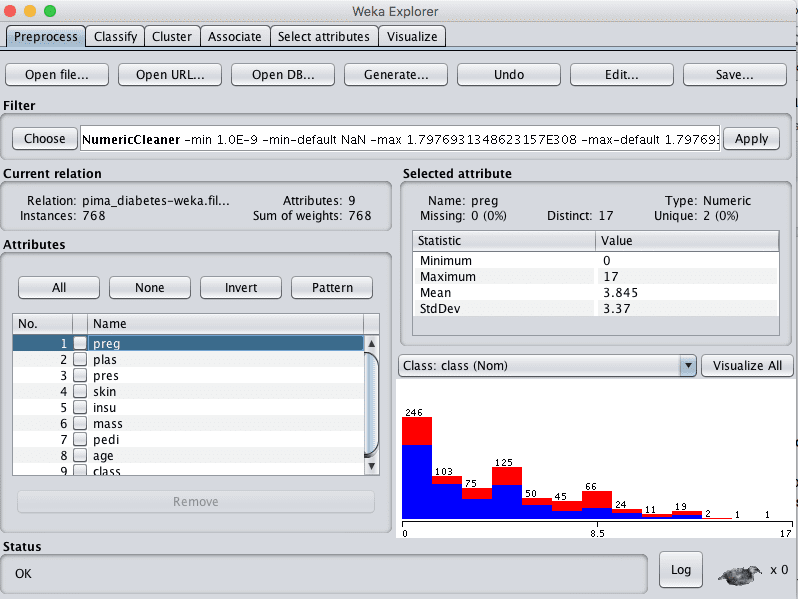
Weka Pima 印第安人数据集数值清理器数据过滤器
现在,让我们用平均值来插补缺失值。
- 在“Filter”窗格中单击“Choose”按钮,然后选择 unsupervised.attribute.ReplaceMissingValues 过滤器。
- 单击“Apply”按钮将过滤器应用于您的数据集。
- 单击“Attributes”窗格中的每个属性,并查看每个属性的缺失值数量。您应该会看到所有属性都没有缺失值,并且属性 2 到 6 的分布应该有所变化。
- 单击“Save…”按钮,导航到合适的目录,然后输入此转换后数据集的合适名称,例如“diabetes-missing.arff”。
- 关闭 Weka Explorer。
您可能还想考虑的其他数据视图包括通过特征选择方法选择的特征子集,以及重新平衡类别属性的视图。
4. 评估算法
让我们设计一个实验,在我们在前面创建的不同数据集视图上评估一系列标准的分类算法。
1. 单击 Weka GUI Chooser 上的“Experimenter”按钮以启动 Weka 实验环境。
2. 单击“New”开始新实验。
3. 在“Datasets”窗格中单击“Add new…”并选择以下 4 个数据集
- data/diabetes.arff(原始数据集)
- diabetes-normalized.arff
- diabetes-standardized.arff
- diabetes-missing.arff
4. 在“Algorithms”窗格中单击“Add new…”并添加以下 8 种多类分类算法
- rules.ZeroR
- bayes.NaiveBayes
- functions.Logistic
- functions.SMO
- lazy.IBk
- rules.PART
- trees.REPTree
- trees.J48
5. 在算法列表中选择 IBK,然后单击“Edit selected…”按钮。
6. 将“KNN”从“1”更改为“3”,然后单击“OK”按钮保存设置。
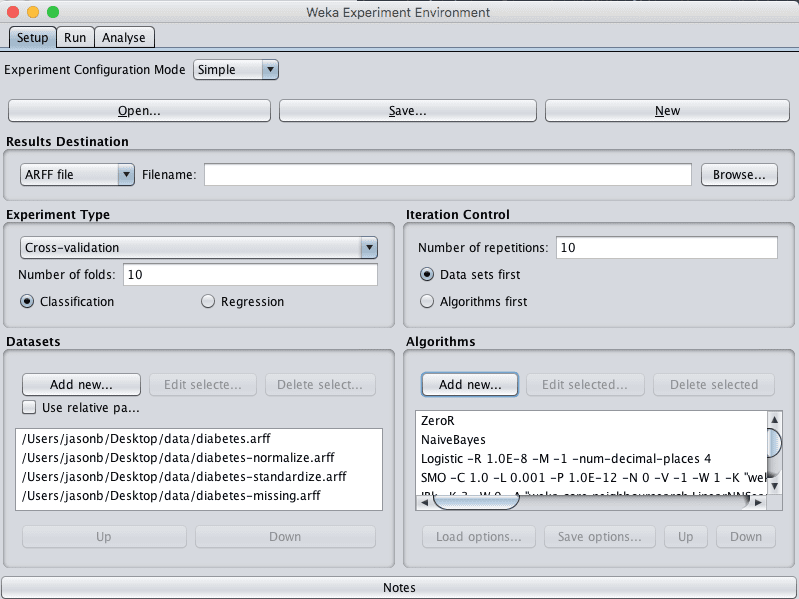
Weka Pima 印第安人数据集算法比较实验
7. 单击“Run”打开 Run 选项卡,然后单击“Start”按钮运行实验。实验应该在几秒钟内完成。
8. 单击“Analyse”打开 Analyse 选项卡。单击“Experiment”按钮加载实验结果。
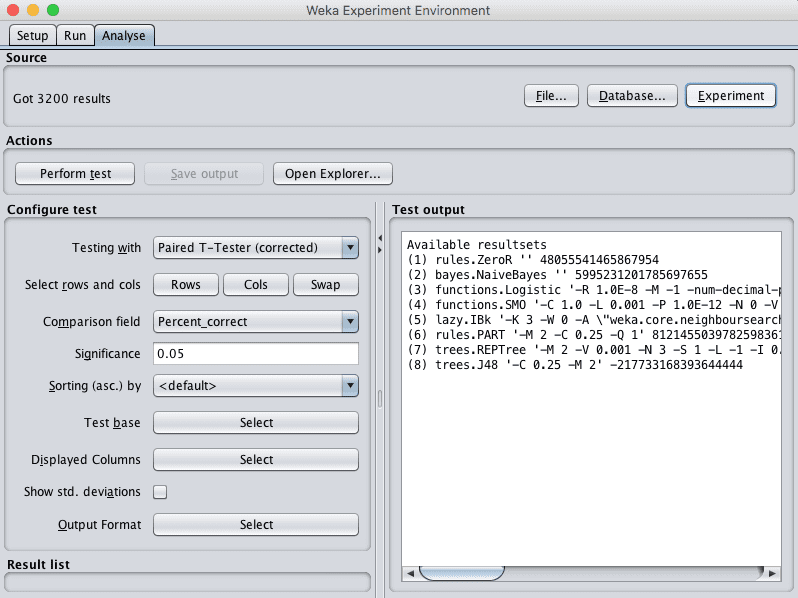
Weka 加载 Pima 印第安人数据集算法比较实验结果
9. 单击“Perform test”按钮,对所有结果与 ZeroR 的结果进行成对 t 检验。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 |
Tester: weka.experiment.PairedCorrectedTTester -G 4,5,6 -D 1 -R 2 -S 0.05 -result-matrix "weka.experiment.ResultMatrixPlainText -mean-prec 2 -stddev-prec 2 -col-name-width 0 -row-name-width 25 -mean-width 0 -stddev-width 0 -sig-width 0 -count-width 5 -print-col-names -print-row-names -enum-col-names" 分析: Percent_correct Datasets: 4 Resultsets: 8 置信度: 0.05 (双尾) 排序方式: - Date: 10/06/16 9:37 AM Dataset (1) rules.Ze | (2) bayes (3) funct (4) funct (5) lazy. (6) rules (7) trees (8) trees -------------------------------------------------------------------------------------------------------------- pima_diabetes (100) 65.11 | 75.75 v 77.47 v 76.80 v 73.86 v 73.45 v 74.46 v 74.49 v pima_diabetes-weka.filter(100) 65.11 | 75.77 v 77.47 v 76.80 v 73.86 v 73.45 v 74.42 v 74.49 v pima_diabetes-weka.filter(100) 65.11 | 75.65 v 77.47 v 76.81 v 73.86 v 73.45 v 74.39 v 74.49 v pima_diabetes-weka.filter(100) 65.11 | 74.81 v 76.86 v 76.30 v 73.54 v 73.03 v 73.70 v 74.69 v -------------------------------------------------------------------------------------------------------------- (v/ /*) | (4/0/0) (4/0/0) (4/0/0) (4/0/0) (4/0/0) (4/0/0) (4/0/0) 键 (1) rules.ZeroR '' 48055541465867954 (2) bayes.NaiveBayes '' 5995231201785697655 (3) functions.Logistic '-R 1.0E-8 -M -1 -num-decimal-places 4' 3932117032546553727 (4) functions.SMO '-C 1.0 -L 0.001 -P 1.0E-12 -N 0 -V -1 -W 1 -K \"functions.supportVector.PolyKernel -E 1.0 -C 250007\" -calibrator \"functions.Logistic -R 1.0E-8 -M -1 -num-decimal-places 4\"' -6585883636378691736 (5) lazy.IBk '-K 3 -W 0 -A \"weka.core.neighboursearch.LinearNNSearch -A \\\"weka.core.EuclideanDistance -R first-last\\\"\"' -3080186098777067172 (6) rules.PART '-M 2 -C 0.25 -Q 1' 8121455039782598361 (7) trees.REPTree '-M 2 -V 0.001 -N 3 -S 1 -L -1 -I 0.0' -9216785998198681299 (8) trees.J48 '-C 0.25 -M 2' -217733168393644444 |
我们可以看到,与 ZeroR 相比,所有算法在所有数据集视图上都表现出一定的技能。我们还可以看到,我们的技能基准是 65.11% 的准确率。
仅从原始分类准确率来看,我们可以看到,将缺失值插补后的数据集视图普遍导致模型准确率较低。此外,标准化和归一化结果与原始结果相比,除了几个百分点的差异外,几乎没有区别。这表明我们可以坚持使用原始数据集。
最后,看起来逻辑回归可能比其他算法取得了更高的准确率结果,让我们检查一下差异是否显著。
10. 选择“Test base”的“Select”按钮,然后选择“functions.Logistic”。单击“Perform test”按钮重新运行分析。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 |
Tester: weka.experiment.PairedCorrectedTTester -G 4,5,6 -D 1 -R 2 -S 0.05 -result-matrix "weka.experiment.ResultMatrixPlainText -mean-prec 2 -stddev-prec 2 -col-name-width 0 -row-name-width 25 -mean-width 2 -stddev-width 2 -sig-width 1 -count-width 5 -print-col-names -print-row-names -enum-col-names" 分析: Percent_correct Datasets: 4 Resultsets: 8 置信度: 0.05 (双尾) 排序方式: - Date: 10/06/16 9:45 AM Dataset (3) function | (1) rules (2) bayes (4) funct (5) lazy. (6) rules (7) trees (8) trees -------------------------------------------------------------------------------------------------------------- pima_diabetes (100) 77.47 | 65.11 * 75.75 76.80 73.86 * 73.45 * 74.46 * 74.49 pima_diabetes-weka.filter(100) 77.47 | 65.11 * 75.77 76.80 73.86 * 73.45 * 74.42 * 74.49 pima_diabetes-weka.filter(100) 77.47 | 65.11 * 75.65 76.81 73.86 * 73.45 * 74.39 * 74.49 pima_diabetes-weka.filter(100) 76.86 | 65.11 * 74.81 76.30 73.54 * 73.03 * 73.70 * 74.69 -------------------------------------------------------------------------------------------------------------- (v/ /*) | (0/0/4) (0/4/0) (0/4/0) (0/0/4) (0/0/4) (0/0/4) (0/4/0) 键 (1) rules.ZeroR '' 48055541465867954 (2) bayes.NaiveBayes '' 5995231201785697655 (3) functions.Logistic '-R 1.0E-8 -M -1 -num-decimal-places 4' 3932117032546553727 (4) functions.SMO '-C 1.0 -L 0.001 -P 1.0E-12 -N 0 -V -1 -W 1 -K \"functions.supportVector.PolyKernel -E 1.0 -C 250007\" -calibrator \"functions.Logistic -R 1.0E-8 -M -1 -num-decimal-places 4\"' -6585883636378691736 (5) lazy.IBk '-K 3 -W 0 -A \"weka.core.neighboursearch.LinearNNSearch -A \\\"weka.core.EuclideanDistance -R first-last\\\"\"' -3080186098777067172 (6) rules.PART '-M 2 -C 0.25 -Q 1' 8121455039782598361 (7) trees.REPTree '-M 2 -V 0.001 -N 3 -S 1 -L -1 -I 0.0' -9216785998198681299 (8) trees.J48 '-C 0.25 -M 2' -217733168393644444 |
看起来逻辑回归的结果确实比其他一些结果(如 IBk、PART、REPTree 和 ZeroR)要好,但与 NaiveBayes、SMO 或 J48 在统计上没有显著差异。
11. 勾选“Show std. deviations”以显示标准差。
12. 选择“Displayed Columns”的“Select”按钮,然后选择“functions.Logistic”,单击“Select”以接受选择。这将仅显示逻辑回归算法的结果。
13. 单击“Perform test”以重新运行分析。
现在我们有了一个最终结果,可以用来描述我们的模型。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 |
Tester: weka.experiment.PairedCorrectedTTester -G 4,5,6 -D 1 -R 2 -S 0.05 -V -result-matrix "weka.experiment.ResultMatrixPlainText -mean-prec 2 -stddev-prec 2 -col-name-width 0 -row-name-width 25 -mean-width 2 -stddev-width 2 -sig-width 1 -count-width 5 -show-stddev -print-col-names -print-row-names -enum-col-names" 分析: Percent_correct Datasets: 4 Resultsets: 8 置信度: 0.05 (双尾) 排序方式: - Date: 10/06/16 9:48 AM Dataset (3) functions.Logist ---------------------------------------------- pima_diabetes (100) 77.47(4.39) | pima_diabetes-weka.filter(100) 77.47(4.39) | pima_diabetes-weka.filter(100) 77.47(4.39) | pima_diabetes-weka.filter(100) 76.86(4.90) | ---------------------------------------------- (v/ /*) | 键 (3) functions.Logistic '-R 1.0E-8 -M -1 -num-decimal-places 4' 3932117032546553727 |
我们可以看到,模型在未见过的数据上的估计准确率为 77.47%,标准差为 4.39%。
14. 关闭 Weka 实验环境。
5. 最终确定模型并呈现结果
我们可以创建模型的最终版本,该模型在所有训练数据上进行训练,并将其保存到文件中。
- 打开 Weka Explorer 并加载 data/diabetes.arff 数据集。
- 单击 Classify。
- 选择 functions.Logistic 算法。
- 将“Test options”从“Cross Validation”更改为“Use training set”。
- 单击“Start”按钮创建最终模型。
- 右键单击“Result list”中的结果项,然后选择“Save model”。选择一个合适的地点并输入一个合适的名称,例如“diabetes-logistic”。
然后可以在以后加载此模型,并用于对新数据进行预测。
我们可以使用上一节收集的模型准确率的均值和标准差来量化模型在未见过的数据上的估计准确率的预期变异性。
我们可以普遍预期,模型在未见过的数据上的性能为 77.47% 加上或减去 (2 * 4.39)% 或 8.78%。我们可以将其重述为准确率在 68.96% 到 86.25% 之间。
这个最终的模型准确率陈述令人惊讶的是,在较低的范围内,该模型仅比 ZeroR 模型略好,ZeroR 模型通过预测所有预测为阴性结果获得了 65.11% 的准确率。
总结
在本贴中,您使用 Weka 机器学习工作台端到端地完成了二元分类机器学习项目。
具体来说,你学到了:
- 如何分析数据集并建议可能有效的数据转换和建模技术。
- 如何设计和保存数据集的多个视图,并在这些视图上抽查多种算法。
- 如何最终确定模型以对新数据进行预测,并呈现模型在未见过数据上的估计准确率。
您对在 Weka 中运行机器学习项目或对此帖有任何疑问吗?请在评论中提问,我会尽力回答。







数据集链接
http://storm.cis.fordham.edu/~gweiss/data-mining/weka-data/diabetes.arff
谢谢。
谢谢 Jason。我学到了很多,我正在开始您的 14 天课程。
谢谢 Hameed,我很高兴听到这个。
嗨,Jason
我喜欢完成本教程。为什么您要设置某些算法的参数?
很高兴听到这个消息。
具体是哪一个?
嗨,Jason,
不错的系列文章。
将一些属性转换为更接近正态分布是否有意义?
可能吧。
你好 Jason,
我是机器学习新手,实际上我想将机器学习用于故障预测和诊断。非常感谢任何课程或指导。
谢谢,欢迎!
我推荐这个通用流程
https://machinelearning.org.cn/start-here/#process