选择数据中合适的特征,可以区分出性能平庸、训练时间长和性能优异、训练时间短的机器学习模型。
Caret R 包提供了工具,可以自动报告数据中属性的相关性和重要性,甚至为您选择最重要的特征。
在本文中,您将通过 R 中的独立示例,了解 Caret R 包中的 特征选择 工具。
阅读本文后,您将了解
- 如何从数据集中删除冗余特征。
- 如何根据重要性对数据集中特征进行排名。
- 如何使用递归特征消除方法从数据集中选择特征。
为您的项目打下基础,阅读我的新书《机器学习实战 (R语言版)》,其中包含分步教程和所有示例的R源代码文件。
让我们开始吧。

机器学习的置信区间
图片由 Paul Balfe 提供,保留部分权利。
需要更多关于R机器学习的帮助吗?
参加我为期14天的免费电子邮件课程,了解如何在您的项目中使用R(附带示例代码)。
点击注册,同时获得该课程的免费PDF电子书版本。
删除冗余特征
数据可能包含高度相关的属性。如果移除高度相关的属性,许多方法将表现得更好。
Caret R 包提供了 findCorrelation 函数,该函数将分析您数据属性的 相关性 矩阵,并报告可以移除的属性。
以下示例加载了 Pima Indians Diabetes 数据集,该数据集包含来自医学报告的许多生物学属性。从这些属性创建了相关性矩阵,并识别了高度相关的属性,在本例中,年龄属性被移除,因为它与怀孕属性高度相关。
通常,您希望移除绝对相关性为 0.75 或更高的属性。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 |
# 确保结果可重复 set.seed(7) # 加载库 library(mlbench) library(caret) # 加载数据 data(PimaIndiansDiabetes) # 计算相关性矩阵 correlationMatrix <- cor(PimaIndiansDiabetes[,1:8]) # 汇总相关性矩阵 print(correlationMatrix) # 查找高度相关的属性(理想情况下 >0.75) highlyCorrelated <- findCorrelation(correlationMatrix, cutoff=0.5) # 打印高度相关属性的索引 print(highlyCorrelated) |
按重要性排名特征
可以通过构建模型来估计特征的重要性。像决策树这样的方法内置了报告变量重要性的机制。对于其他算法,可以使用对每个属性进行的 ROC 曲线分析来估计重要性。
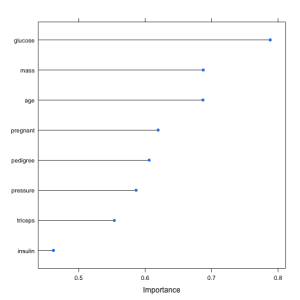
下面的示例加载了 Pima Indians Diabetes 数据集并构建了一个 学习向量量化 (LVQ) 模型。然后使用 varImp 来估计变量重要性,并进行打印和绘图。它显示葡萄糖、体重指数和年龄属性是数据集中最重要的前 3 个属性,而胰岛素属性是最不重要的。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 |
# 确保结果可重复 set.seed(7) # 加载库 library(mlbench) library(caret) # 加载数据集 data(PimaIndiansDiabetes) # 准备训练方案 control <- trainControl(method="repeatedcv", number=10, repeats=3) # 训练模型 model <- train(diabetes~., data=PimaIndiansDiabetes, method="lvq", preProcess="scale", trControl=control) # 估计变量重要性 importance <- varImp(model, scale=FALSE) # 汇总重要性 print(importance) # 绘制重要性图 plot(importance) |
特征选择
可以使用自动特征选择方法来构建具有不同数据集子集的模型,并识别哪些属性是必需的,哪些是不必需的,以构建准确的模型。
Caret R 包提供的一种流行的自动特征选择方法称为 递归特征消除 (RFE)。
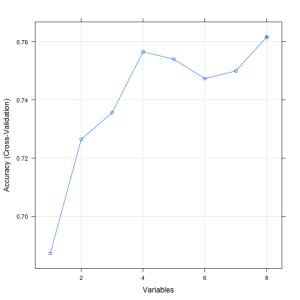
下面的示例提供了在 Pima Indians Diabetes 数据集上使用 RFE 方法的示例。每次迭代都使用随机森林算法来评估模型。该算法配置为探索所有属性的可能子集。在此示例中选择了所有 8 个属性,尽管在显示不同属性子集大小的准确性图表中,我们可以看到仅使用 4 个属性就能获得几乎可比的结果。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 |
# 确保结果可重复 set.seed(7) # 加载库 library(mlbench) library(caret) # 加载数据 data(PimaIndiansDiabetes) # 使用随机森林选择函数定义控制 control <- rfeControl(functions=rfFuncs, method="cv", number=10) # 运行 RFE 算法 results <- rfe(PimaIndiansDiabetes[,1:8], PimaIndiansDiabetes[,9], sizes=c(1:8), rfeControl=control) # 汇总结果 print(results) # 列出选定的特征 predictors(results) # 绘制结果图 plot(results, type=c("g", "o")) |
总结
在本文中,您了解了 caret R 包提供的 3 种特征选择方法。具体来说,搜索和删除冗余特征,按重要性对特征进行排名,以及自动选择最有预测能力的特征子集。
提供了三个独立的 R 示例,您可以将其复制粘贴到您自己的项目中,并根据您的具体问题进行调整。


感谢这篇很棒的文章。您的写作总是切中要点,讲解实用知识,我非常喜欢,易于理解!我认为您在递归特征消除或 RFE 中遗漏了一点。需要包含库 “library(randomForest)”,因为当我尝试复制时,无法找到 Random Forest 包。
继续写作,它非常有帮助!!!🙂
谢谢。
你好,
我尝试运行上面帖子中的特征重要性,但失败了。
我收到一个关于包 ‘e1071’ 的错误。我用来安装它的命令是
install.packages(“e1071”,dep=TRUE,type=’source’)
错误是:
ERROR: compilation failed for package ‘e1071’
我正在使用 R 3.1.1 的最新版本。
有人能帮帮我吗?
> install.packages(‘e1071’)
正在尝试 URL ‘https://cran.hafro.is/bin/windows/contrib/3.6/e1071_1.7-2.zip’
内容类型 ‘application/zip’ 长度 1021699 字节 (997 KB)
已下载 997 KB
包 ‘e1071’ 已成功解压并完成了 MD5 校验
已下载的二进制包位于
太棒了!
这是一篇很棒的文章!谢谢!我有一个问题。对于我的数据集,我执行了最后两个选项(按重要性排名,然后进行特征选择),但是这两种方法选择的顶级特征并不相同。为什么会这样?哪种方法更好?
谢谢!
谢谢 Jordan。
这是一个好问题。不同的方法会选择不同的特征子集。就像没有最佳模型一样,很可能也没有“最佳特征集”。我的建议是尝试使用每个特征子集进行建模,看看哪种最适合您的问题和需求。
应用机器学习是一个实证假设检验的过程——大量的试错。
感谢如此有用的帖子。
我有一个关于 RFE 方法的问题。我不确定我是否正确理解了这一点,因为我想知道在每个步骤中使用了哪种方法来构建模型,以及是否有可能在每次迭代中使用 SVM 来构建模型?
您的帖子写得很好——我对 caret 还不熟悉,发现它有助于快速运行。我正在处理一个 p>>n 分类问题,特别是,我并不关心一个黑箱预测模型,而是一个更具解释性的模型,因此我正在尝试提取能够帮助解释结果的重要特征集(我有额外的数据来验证提取的特征之间的关系)。
我想知道使用不同的模型运行 RFE 并提取拟合每个模型的重要变量子集,然后找到特征集的交集是否有意义?我预计每个模型可能会产生不同的特征集(其中最重要的特征会共享),并且一个模型的特征集可能不适用于另一种类型的模型,所以我不太确定。
如何最好地处理这样的问题?谢谢!
精彩的帖子,而且排版非常好。非常感谢。
可以使用 caret 包进行特征提取吗?
很棒的图片,但在运行代码时,我发现 RF 特征选择会推荐 5 个特征作为推荐,如何更改默认设置?非常感谢!!!
我有同样的问题。如何绘制所有变量而不是前 5 个?
我有一个关于机器学习特征选择方法的问题。您使用了整个数据集,是否需要将其拆分为训练集和测试集?然后构建一个模型用于训练集,并在此查看 VarImp?
您好。很棒的帖子!我有一个关于使用 varImp 进行特征选择的疑问。我编写了一个算法,该算法运行 randomForest 来构建训练集上的模型。现在我想在应用 randomForest 之前使用 varImp() 来仅选择重要的特征。但是 varImp 似乎又要构建一个模型来提取特征重要性,这似乎有些适得其反。您能否向我解释一下在使用 randomForest 训练训练数据模型之前使用 varImp 的意义?在这种情况下,我该如何使用 varImp?
感谢这篇信息丰富的帖子。
我正在使用 Lending Club 网站上公开提供的数据。该数据集包含大量非数值列(grade、loan status 等)。我假设在我进行上面描述的特征选择方法之前,我需要将这些非数值数据映射到数值。我的问题是:处理这种情况是否有规定方法,还是可以遵循临时映射方案?当然,我的目的是能够进行智能的特征选择。
Amit
你好,James,
我关注您的新闻通讯。它们在关键时刻拯救了我两次!谢谢。另外,这些算法中有哪些会考虑数据标准化吗?
感谢这些有用的信息。
如何将其应用于 SVM?
非常感谢这些精彩的信息!
似乎特征重要性仅适用于 LVQ 方法进行分类问题,而不适用于回归问题,对吗?
Jason,我遇到的最棒的 ML 帖子!!
我尝试了代码,并尝试仅为 RFE 算法选择的特征建模。当我尝试使用时出现错误。无法弄清楚为什么会给出错误
‘Error in eval(expr, envir, enclos) : object ‘diabetes’ not found’ 在 model (….) 语句。
#加载库
rm(list=ls())
library(mlbench)
library(caret)
#加载数据
data(PimaIndiansDiabetes)
#使用随机森林选择函数定义控制
control <- rfeControl(functions=rfFuncs, method='cv', number=10)
#运行 RFE 算法
results <- rfe(PimaIndiansDiabetes[,1:8], PimaIndiansDiabetes[,9], sizes=c(1:8), rfeControl = control)
#汇总结果
print(results)
names(PimaIndiansDiabetes)
#列出选定的特征
predictors(results)
#绘制结果
plot(results, type=c('g','o'))
PimaIndiansDiabetes$diabetes <- as.character(PimaIndiansDiabetes$diabetes)
PimaIndiansDiabetes$diabetes[PimaIndiansDiabetes$diabetes=='neg'] <- 0
PimaIndiansDiabetes$diabetes[PimaIndiansDiabetes$diabetes=='pos'] <- 1
PimaIndiansDiabetes$diabetes <- as.factor(PimaIndiansDiabetes$diabetes)
View(PimaIndiansDiabetes)
#拆分数据
inTrain = createDataPartition(y=PimaIndiansDiabetes$diabetes, p=0.7, list=FALSE)
train <- iris[inTrain,]
test <- iris[-inTrain,]
attach(train)
#训练模型
model <- train(diabetes ~ glucose + mass + age + pregnant + pedigree, data=train, trControl = train_control, method='lvq', tuneLength=5)
#模型摘要
summary(model)
您需要使用 RStudio 中的“PimaIndiansDiabetes”数据集,而不是此页面上提供的(pima.indians.diabetes.data)。
嘿,我正在尝试绘制不同变量的重要性,但是调用
importance <- varImp(model, scale=FALSE)
会产生错误
Error: (list) object cannot be coerced to type 'double'
有什么建议告诉我哪里出错了或者如何调试这个问题吗?
提前感谢!
很棒的帖子 Jason。我一直在像大学课程一样关注您的博客,以便尽快掌握 ML。
在重要性部分,例如在您的例子中,变量‘Age’排名第三,但我能否知道它是以积极还是消极的方式?也就是说,年龄越大导致患糖尿病的几率越大,还是反之?
所以,我像这样进行了特征消除
control <- rfeControl(functions=caretFuncs, method="cv", number=10)
results <- rfe(mydata.train[,1:23], mydata.train[,24], sizes=c(2,5,8,13,19), rfeControl=control , method="svmRadial")
print(results)
递归特征选择
外部重采样方法:交叉验证(10 折)
子集大小的重采样性能
变量 准确率 Kappa 准确率SD KappaSD 选择
2 0.5100 -0.02879 0.05230 0.08438
5 0.5371 0.02703 0.05953 0.12621
8 0.5371 0.03630 0.07200 0.15233
13 0.5207 0.01543 0.05248 0.11149
19 0.5850 0.15647 0.07122 0.14019
23 0.6447 0.27620 0.06088 0.12219 *
这对我是个好结果,准确率约为 65%。但是当我尝试使用
进行训练时
svm.model <- train(OUTPUT~.,data = mydata.train,method = "svmRadial",trControl = trainControl(method = "cv",number = 10),tuneLength = 8,metric="Accuracy")
,我无法达到那个准确率。递归消除使用的是什么模型和参数?
嗨!
这篇博客信息量很大。谢谢!
很高兴能看到一些推荐系统示例,例如超市购物篮分析以及对 R 中的用户的推荐。
它的工作原理。
您能否为此添加一个页面?
谢谢!
很棒的帖子!我尝试使用我的数据集 R_feature_selection_test.csv
但它不起作用。我收到如下错误消息。您能给我一些建议吗?
data(R_feature_selection_test)
警告信息
在 data(R_feature_selection_test) 中
数据集 ‘R_feature_selection_test’ 未找到
我遇到了同样的问题。您找到解决方案了吗?
这是一篇旧帖子,但以防万一有人需要
您需要先将数据导入 R。
****
mydata <- read.csv("R_feature_selection_test.csv")
***
感谢分享!
您好,感谢您的精彩博客。我的因变量被编码为疾病缺席 = 0,疾病存在 = 0。输入包含二元和连续变量。二元变量被编码为 0 和 1。
当我尝试 lvq 时,我收到以下错误消息:
Error in train.default(x, y, weights = w, …)
回归模型类型错误
稍后当我尝试 rfe 时,我收到以下警告:
In randomForest.default(x, y, importance = first, …)
响应有五个或更少个唯一值。您确定要进行回归吗?
您知道是什么原因造成的吗?
我猜问题是:caret 中的 lvq 和 ref 是否适用于分类数据?有人将这些模型应用于包含分类变量的数据集吗?
当我将输出重新编码为“yes”、“no”而不是 1/0 时,这两行现在都可以工作了。请确保在重新编码的输出存储为字符格式时将其设置为 as.factor。
太棒了!谢谢
您好,我也遇到了类似的问题,但我的因变量不是“yes”、“no”。它是一些数字。我也可以用它来进行特征选择吗?
是的。您可能需要将因子编码为 0 和 1。
嗨 Jason
非常好的工作。
我想问一下我们如何使用t检验进行特征选择。我有12个属性(变量)和一个类别变量(标签)。请您指导我,我该如何使用t检验进行变量选择?
谢谢,
只是想感谢您写了这篇易于理解的帖子!
非常欢迎。
嗨,Jason,
感谢您提供的代码具有极高的可复现性。它对我来说效果很好,我也能将其应用于我当前的问题。既然您最近回复了一个帖子,我希望能请您解答一个关于RFE非常基础和普遍的疑问。
我一直认为RFE是用于补充caret包的train函数或random forest包的randomForest函数训练模型的额外工具,直到我最近读到一篇论文,它没有明确说明,但暗示特征选择是在训练随机森林模型之前完成的。那么,哪种情况是RFE的合适用例?
另外,我使用RFE函数得到的准确率与我通过调整ROC模型得到的准确率不同。这可能是原因吗?
非常感谢您的时间,
祝好,
Manasi
通常,您可以先进行特征选择,然后构建模型。
作为特征选择的一部分,您可以构建模型,但仅用于告知您选择哪些特征。这些不是最终模型。
谢谢,这证实了我的疑虑,并有助于我进一步将此方法纳入分析中。
祝好,
Manasi
很高兴听到这个消息,Manasi。
亲爱的 Jason,
您能否使用SVM-RFE,然后是遗传算法,然后是排列测试,最后是R代码中的任何其他模型来构建一个特征选择模型?
抱歉,Krishna,我没有能力为您编写此代码。
祝你的项目一切顺利。
嗨 Jason
感谢您提供的详细说明。这对像我这样的新人非常有帮助。我有一个疑问,在使用上述技术之前,我们是否需要去除异常值?
是的,数据清理通常是一个好的第一步。
很棒的帖子!我尝试使用我的数据集 R_feature_selection_test.csv
但它不起作用。我收到如下错误消息。您能给我一些建议吗?
data(R_feature_selection_test)
警告信息
在 data(R_feature_selection_test) 中
数据集 ‘R_feature_selection_test’ 未找到
你好Jason,很棒的帖子!
我的原始数据集中某些列存在缺失值,但为了使用rfe(),我需要处理这些缺失值。如果我处理了缺失值,我的特征选择将基于此,但在最终模型中,我没有为那些列处理缺失值,我的结果会不会有偏差?
有可能性。
尝试使用经过插补缺失值的数据进行特征选择,然后尝试使用删除了所有缺失值的记录进行特征选择。
看看哪组特征能使模型达到最佳性能。
希望这能有所帮助。
好的,谢谢!
另外,有没有办法决定算法的迭代次数,还是我们只需尝试不同的次数,然后尝试找出最佳次数?
尝试不同的训练长度,看看哪种效果最好。
嗨!
我对编程这件事是新手。
我试图根据重要性对特征进行排名,但一直收到错误:Error in na.fail.default(list(SalePrice = c(208500L, 181500L, 223500L,
对象中有缺失值
我的代码看起来像这样
model <- train(SalePrice~., data=train_data, method="lvq", preProcess="scale", trControl=control)
其中train_date是我的数据框的名称,其中包含我所有的数据。您知道为什么我一直收到这个错误吗?
Brittany,我没有见过这个特定的错误。我想这可能是您数据的问题。
也许尝试缩减您的数据,无论是行还是列,直到您的代码开始工作——这可能有助于找出原因。
你好Brittany/Jason,
您最终找到这个问题的解决方案了吗?抱歉带来任何麻烦。
嗨 Jason
这是一篇很棒的帖子,谢谢。
我正在尝试在包含约1000个数据条目和17个变量的数据集上运行RFE,其中几个变量是分类的。我没有收到错误,但是,该过程似乎一直在运行,没有停止也没有得出任何结论。
您知道这是否可能与我的数据集大小或数据类型有关?
非常感谢
Ciara
你好Ciara,RFE可能不是分类输入的最佳方法。
尝试包装一个树模型,看看效果如何。
更多关于特征选择的信息请看这里
https://machinelearning.org.cn/an-introduction-to-feature-selection/
Jason你好
感谢这篇恰到好处的帖子。
然而,我遇到了同样的问题(处理不会结束),而且我的变量不是分类的。
可能是什么问题?
也许将数据缩减到一小部分,以帮助找出故障原因?
你好Jason,感谢您的精彩解释。我有一些关于交叉验证的问题。
1.我们为什么要使用交叉验证?它只是为了选择重要的特征吗?还是有其他原因?
2.我们什么时候做交叉验证?是在模型构建之前还是之后?
希望能得到回复。
谢谢
Gaurav
你好Gaurav,
交叉验证使我们能够通过估计选择结果在未见过的数据上的表现来做出决策(选择模型或选择特征)。这比仅回顾整个训练数据集的表现更稳健。
它用于选择要构建的模型,是事先进行的。一旦选定,就可以使用所有可用数据来构建模型。
希望这能有所帮助。
你好,
您能否解释一下如何在R中使用遗传算法对PimaIndiansDiabetes数据集进行特征选择?如果可能的话,在SAS中也请。
抱歉,我还没有使用遗传算法进行特征选择的示例。至少目前还没有。
感谢分享您的知识,Jason。精彩的解释。
不客气,Roberto,很高兴您觉得它有用。
老师好,是否有针对二进制分类的遗传算法特征选择的教程?我正在处理DNA和RNA数据集。
抱歉,Asad,目前还没有关于GA用于特征选择的教程,希望很快能有。
很棒的帖子Jason。
我尝试固定ntree并找到不同的mtry。
代码工作正常,但不同mtry值的重采样具有相同的准确度范围。不可能...有些东西不起作用!!!
谢谢您的回复
metric <- "Accuracy"
control <- trainControl(method="cv", number=10, search="grid")
tunegrid <- expand.grid(mtry=c(10, 20, 30, 40, 50))
modellist2 <- list()
for (mtry in c(10, 20,30, 40, 50)) {
set.seed(seed)
custom2 <- train(Class~., data=dataset, method="rf", metric=metric, tuneGrid=tunegrid, trControl=control, ntree=500)
key2 <- toString(mtry)
modellist2[[key2]] <- custom2
}
custom2
results2 <- resamples(modellist2)
summary(results2$values)
好信息!
我有两个问题。
1. 在varImp选择中,当我执行该代码时,我可以看到一个总体。
什么是总体??(例如p值、t检验……那是什么?)
2. 在varImp()结果中,应该选择或删除哪些变量??标准是什么??
您可以在官方文档中了解有关该功能的更多信息:
https://topepo.github.io/caret/variable-importance.html
您的帖子真的很有帮助,非常感谢这些信息,它只适用于定量变量,我需要知道如何使用定性变量计算矩阵。
好问题,抱歉我目前没有示例。
谢谢您的帖子,Jason!有没有适用于名义数据的特征选择的包(算法)?
我认为肯定有,但我一时想不起来,抱歉。
有没有办法让findCorrelation()标记严格高于截止值的属性?从我的用例来看,绝对相关值会与截止值进行比较,如下面的详细输出片段所示(cutoff=0.9)
组合行12474和列12484高于截止值,值为0.922
标记列12474
组合行12476和列12484高于截止值,值为-0.913
标记列12476
有没有办法不标记负相关?
谢谢你的帮助!
它可能在绝对值 abs() 上工作。
嗨,Jason,
我假设在开始使用上述特征选择方法之前,我需要将这些非数字值转换为数字值(例如,将“W”表示获胜转换为“1”,将“L”表示失利转换为“0”)。这是对的吗?或者,我可以将那些带有非数字值的列保留原样吗?我的意图当然是能够进行智能的特征选择。
提前感谢。
李
通常是的,大多数方法都期望处理数值。
嗨,Jason,
感谢您写了这篇很棒的帖子!我想知道如何将相同的技术应用于大型数据集,以便保留所有特征并提取高度相关的所需行(作为子集或样本)。假设我们从一个包含100万行和15个变量的矩阵开始,我想提取20行相关性最高或最低的行。哪种技术(或算法)最好?提前感谢!
Mike,这是一个有趣的想法。您可能需要为此任务准备一些自定义代码。
嗨,Jason,
非常感谢您的解释。现在我正在尝试将其应用于另一个数据集,但遇到了一些问题。我的数据集有962个特征和144个观测值。我的响应是一个有4个级别的因子,所有其他变量都是数值或整数。到目前为止,我能够运行第一部分,但在构建模型时出现了一个错误。
library(mlbench)
library(caret)
set.seed(233)
correlationMatrix <- cor(dataset[,3:962])
highlyCorrelated <- findCorrelation(correlationMatrix, cutoff = 0.5)
control <- trainControl(method = "repeatedcv", number=10, repeats=3)
model Error in seeds[[num_rs + 1L]] : subscript out of bounds
这是我第一次遇到这个错误,我还没有找到任何能帮助我的信息。我想知道您是否有什么提示?
谢谢!
很遗憾听到这个。我没有见过这个错误。
也许尝试简化您的代码/运行以帮助暴露它。
也许可以尝试在stackoverflow/crossvalidated上搜索,甚至在那里发布问题?
遇到同样的问题了吗?您解决了它吗?
我遇到了同样的问题。您解决了它吗?
你好Jason!我正在使用特征选择,复制并调整代码以适应我的数据,而不是根据ACCURACY给出结果,它根据RMSE给出结果。如果我在多元线性回归中使用选定的变量,这会导致不同的RMSE值。您能否帮我将此图从RMSE更改为准确性?谢谢!
如果您的问题是回归问题(预测一个实数值),那么您无法计算准确性,您必须计算预测误差,例如RMSE。
你好Jason,感谢您分享这些精彩的帖子。
我有一个问题:机器学习算法的特征数量与观测数量之间是否存在任何限制?例如,我能否将更多的特征与观测值一起运行SVM或随机森林?例如,对于线性回归,我读到(作为经验法则)特征数量最好不要超过观测数量的1/5,以避免过拟合。
谢谢!
一般来说,我们更希望观测数量多于特征数量。事实上,也许是10倍或更多的观测值比特征多。
获取尽可能多的数据是最好的经验法则。
https://machinelearning.org.cn/much-training-data-required-machine-learning/
感谢您分享的精彩帖子!我喜欢像这样简单的代码的简单解决方案。这些帖子对我的项目非常有用。非常感谢!Piroska
谢谢Piroska,很高兴听到这个!
嗨 Jason
感谢您的帖子。
在您的帖子中,您使用了一个数字数据集。
是否可以将上述方法应用于混合数据集,例如UCI的心脏病和
肝炎数据集?
谢谢
也许可以,但您需要将分类变量编码为整数或二进制向量。
如何使用ROC曲线分析获得每个属性的重要性?
为什么findCorrelation只报告第一行?如何让它报告所有相关性?
您可以通过输入 ?cor 来了解更多关于 cor 函数的信息。
帮助我遇到了这个错误 results <- rfe(data[,1:71], data[,72], sizes=c(1:71), rfeControl=control)
Error in { : task 9 failed – "Can't have empty classes in y."
仔细检查您的数据,例如,将其分配给一个变量并对其进行汇总,以确保它符合您的预期。
model <- train(diabetes~., data=PimaIndiansDiabetes, method="lvq", preProcess="scale", trCo
您能解释一下diabetes指的是什么吗?或者它来自哪里?
数据集(PimaIndiansDiabetes)中的结果变量(列)。
嗨,
我想理解您的代码,您能帮我吗?
model <- train(diabetes~., data=PimaIndiansDiabetes, method="lvq", preProcess="scale", trControl=control)
在上述语句中,diabetes指的是什么?
当然,‘diabetes’是‘PimaIndiansDiabetes’数据集中的目标列。
抱歉,忽略我最后的帖子
你好,
我正在尝试运行您的代码,替换我的数据集,但它给了我一个关于特征选择的Varimp的回归错误模型。
有什么想法可能导致此问题?
也许把你的错误发到 stackoverflow 上?
我尝试过了,但没有得到任何回应。
嗨,我正在尝试,但它说没有名为caret的包,也没有名为mlbench的包。请帮忙。
很遗憾听到这个。也许尝试在stackoverflow上发布您的错误消息?
如果错误消息是“Error in library(mlbench)”
在此之前,您需要执行此操作
> install.package(“mlbench”)
> 我在安装Fselector命令时遇到了问题,我不明白问题出在哪里
请帮忙
install.packages(“FSelector”)
Error in fetch(key)
lazy-load database ‘C:/Users/ux305/Documents/R/win-library/3.4/FSelector/help/FSelector.rdb’ is corrupt
> library(FSelector)
Error: package or namespace load failed for ‘FSelector’
.onLoad failed in loadNamespace() for ‘rJava’, details
call: fun(libname, pkgname)
error: JAVA_HOME cannot be determined from the Registry
很遗憾听到这个。也许尝试将错误发布到stackoverflow?
我想问一下,在分类情况下,特征选择的最佳性能评估指标是什么(ROC、MSRE、、ACCURACY……)?谢谢。
这真的取决于您的项目、您的目标和您的特定数据集。
你好Jason,这是一篇非常好的帖子,我是一个忠实粉丝,因为您所有的工作都使ML易于处理。只是想知道您的帖子是否更进一步地完成了主要的分类任务,即训练选定的rfe属性?
抱歉,在这个阶段我没有RFE的更多细节。
这真的很好,但当我使用
model <- train(quality~., data=wine, method="lvq", trControl=control)
然后
error: wrong model type for regression.
这个模型是否不适用于仅数值数据?
我正尝试将此模型应用于红酒数据集。
也许可以将葡萄酒质量分数视为10个类别?
嗨 Jason,非常感谢您发表这篇帖子。
不客气。
嗨,Jason;
在特征选择中,当您使用 plot(results, type=c(“g”, “o”)) 代码制作 accuracy vs Variables 图时,是否有办法获得变量的实际名称而不是变量编号?
提前感谢您的帮助。
你好 jason,
我在使用 Caret 包的 RFE-RF 时遇到问题
set.seed(42)
index <- createDataPartition(RFTXModel$outcome, p = 0.7, list = FALSE)
train_data <- RFTXModel[index, ]
test_data <- RFTXModel[-index, ]
x <- dplyr::select(train_data, -outcome)
y <- as.factor(train_data$outcome)
set.seed(42)
control <- rfeControl(functions = rfFuncs,
method = "repeatedcv",
repeats = 5,
verbose = FALSE)
Result <- rfe(x,y,metric = "Kappa",
rfeControl = control)
print(Result)
我有两个问题
1.如果我只需要一个用于另一个分类器的预测变量列表,我是否需要为 RFE 分割训练和测试数据?
2.如何获取每个变量的 OA 和 Kappa 值,如这张表所示
变量名 OA Kappa SD (OA) SD (Kappa)
PC2Tex1 0.63 0.57 0.05 0.06
PC2 0.70 0.65 0.03 0.04
提前感谢
在去除冗余特征时,我们不关注负相关吗?
是的,我们应该。或者直接取系数的绝对值,然后在正域工作。
嗨 Jason。如果我使用递归特征消除,如何获得最佳模型的 ROC 曲线?例如,我有 10 个变量,它选择了 5 个,我如何为这个特定模型绘制曲线?
谢谢!
选择特征后,拟合模型,然后使用测试数据集计算 ROC 曲线。
抱歉,我认为我没有 R 的示例。
嗨,杰森,
我有一个问题。我的数据集有 1600 个特征。我已经应用了 lvq。
它给出了 20 个重要的特征。但我需要至少 60 个特征。如何使用 varimp 获得?plot(importance) 太笨拙了,我无法获得特征的名称。
我不确定,抱歉。也许可以问问 stackoverflow?
嗨 Jason,我可能不小心把它发到了另一个帖子,但我很好奇 caret 包的 Var Imp 图和常规的 Random Forest Var Imp 图有什么区别。在常规 RF 中,变量重要性由 gini 减少决定,而您在此处所说的似乎是 caret 使用了不同的方法 - 而不是 gini 减少。在这两个包中运行的代码相同(VarImp) - 所以我有点困惑……
Caret 实际上并没有实现算法,它只是一个包装器,用于使用其他包(如随机森林包)中的算法。
哦,我明白了,谢谢……但是我们从 caret 随机森林生成的图是基于 1-100 的重要性图,而在单独使用随机森林时,我们得到的是平均准确率下降和平均 gini 下降作为重要性。我想这就是我感到困惑的地方,因为我曾假设 caret 使用的是 RF 包。但是当我同时运行重要性图时,它们似乎没有给我相同的统计数据(即使变量的顺序相同)。
我明白了。也许可以问问 stackoverflow 或 R 用户列表?
感谢这篇帖子!非常有帮助。我按照您的指示运行了模型。它显示我的 21 个变量可以缩小到 8 个。它列出了变量 1-7 和 21。这些数字是否对应于它们在数据框中出现的顺序?提前感谢!
是的,这些数字代表每个选定特征的列索引。
为什么我们要删除相互关联的特征?如果我发现 2 个特征高度相关,我应该删除其中一个还是两个都删除?为什么没有人问这个问题?
它可以简化模型并消除冗余。这意味着它更简单,学习速度更快,甚至可能做出更好的预测。
您删除其中一个。
您删除与因变量相关性较低的那个。
感谢这篇精彩的文章。如果我有一个分类问题,假设我想选择一个算法,我有一个问题。
我先使用 RFE,得到重要特征,然后基于这些特征运行所有可能的算法(例如,逻辑回归、kNN、决策树……)并选择最佳算法吗?
还是我为每个算法选择特征(例如,逻辑回归中的前向选择,决策树中的 RFE 等),然后在最后比较所有算法的准确性并选择最佳算法?
也许可以来回尝试这两种方法,直到找到特征和模型的良好组合。
您能否提供一个关于使用 .75(或其他)截止值的参考或一组参考?
谢谢你
没有,抱歉。
也许可以对不同的截止值进行敏感性分析,看看哪种最适合您的数据集。
感谢分享一个好方法。我有一个问题。如果我想知道具有不同数量变量的变量的详细组合,我该怎么做?
抱歉,我没跟上,您能详细说明一下吗?
例如,我有 11 个变量。当变量数量为 7 时,模型是最优的,我可以知道哪些变量是。我想知道当变量数量为 5 时,具体是哪个变量。
嗨,Jason,
我有一个关于相关性的问题 - 我通过此函数找到的高度相关特征……
# 查找高度相关的属性(理想情况下 >0.75)
highlyCorrelated <- findCorrelation(correlationMatrix, cutoff=0.75)
…我需要删除所有这些还是只删除一半,因为那样它们就不再与其他任何东西相关了。还是说这些特征与我数据集中的所有变量都高度相关?感谢您的帮助。
祝好,
Steven
一半。
目的是消除冗余特征。那些与现有特征高度相关的特征是冗余的,它们不提供新信息。
嗨,Jason!
您是否有关于变量归一化的话题?
是的,有很多,也许可以从这里开始
https://machinelearning.org.cn/?s=normalize&post_type=post&submit=Search
嗨,Jason,
很棒的帖子,非常感谢。
我有一个关于“递归特征消除”的问题。这里显示,使用 4 个特征可以获得与使用 8 个特征几乎相同的性能,有没有办法知道是哪 4 个变量?
非常感谢
是的,它会报告每个选定变量的索引或列。然后您可以查找这些列的名称。
这非常有帮助。它对我的数据效果很好
谢谢,很高兴对您有帮助。
变量重要性和特征选择有什么区别?
重要性可用于选择,但本身不是选择。
您能否告诉我变量的重要性是如何衡量的?它们是平均准确率下降还是 Gini 下降?
谢谢你。
是的,您可以在这里了解更多
https://topepo.github.io/caret/variable-importance.html
嗨,Jason,
如果我使用基于随机森林的 RFE,是否可以将选定的特征集用于构建其他类型的模型,如 SVM?
谢谢!
是的。
您能否分享一下 RFE 的优缺点?我使用过它,也使用了其他示例 xgboost 和 GA 来比较它们的能力。在某些情况下,RFE 表现良好,而在其他情况下,xgboost 和 GA 特征模型获得了更高的准确性。
ps. 使用了回归数据。
此致
如果它在您的数据集上表现良好,就使用它,否则就不要。除此之外,我帮不了您。
嗨
感谢您清晰明了的解释。
如果我想为 caret 包中的其他 ML 算法(例如 SVM、ANN、KNN)进行递归特征选择,该怎么办?
我看到一个名为“caretFuncs”的免费函数。我是否可以将此函数用于 caret 包中嵌入的所有机器学习方法,包括随机森林分类器?提前感谢。
是的,我认为您可以在 RFE 中使用任何您喜欢的算法。
嗨,Jason,
感谢这篇好文章。
我注意到相关矩阵实际上是皮尔逊相关计算。
所以我一直在想,在我们观察到的相关矩阵中,有 4 个特征高度相关。在开始新的预测模型之前,我们是否需要删除这 4 个特征中的 3 个?
谢谢,
此致,
Dominique
是的。
我相信有一个程序可以做到这一点,用于删除冗余输入。据我回忆,我认为每个特征都会根据其与其他所有特征的相关程度进行评分,并移除最相关的输入子集。
嗨,我正在使用以下代码进行递归消除,这是一个 140:396 的数据集。
#递归消除……………………………………
control <- rfeControl(functions=rfFuncs, method="cv", number=10)
RFE <- rfe(dat5[,2:396], dat5[,1],
sizes=c(1:8), rfeControl=control)
但是我一直收到以下错误。
Error in summary.connection(connection) : invalid connection
这是一个奇怪的错误。
也许可以尝试将您的代码和错误消息发布到 R 用户组或 stackoverflow?
感谢回复,Jason,
但错误是由于并行计算引起的。
运行以下代码后,能够解决它
library(doParallel); library(doSNOW);
{ # doParallel
cl <- makeCluster(detectCores()); registerDoParallel(cl);
getDoParWorkers(); stopCluster(cl);
# let it snow (doSNOW)
cl <- makeCluster(32,type="SOCK")
stopCluster(cl)
}
很高兴听到这个消息。
亲爱的 Jason,
感谢您的工作。我有一个关于 varImp() 之后特征选择部分的问题。在看到最重要的预测特征后,假设我想选择前 4 个作为我的预测变量,并继续进行随机森林。您能帮我吗?
一个极好的问题。我希望将来能用一个例子来演示它。
嗨 Jason
关于我定义的基础
sizes = C (1: 8)
用于
results <- rfe(PimaIndiansDiabetes[,1:8], PimaIndiansDiabetes[,9], sizes=c(1:8), rfeControl=control)
我猜这取决于数据集,但有什么一般性的规则可以参考吗?
提前感谢您的回复
不一定,测试 1 到 n(对于 n 个输入特征)可能是一个好主意。
嗨,Jason,
我有一个非线性时间序列数据集,包含数值数据,我只传递模型 <- train(EGT~., data=df[1:10], method="lvq", preProcess="scale", trControl=control)
它会得到错误:错误的回归模型类型。
我不知道。另外,如何计算非线性相关性?!
谢谢。
这种特征选择可能不适用于时间序列。
也许可以尝试非参数相关,比如 Spearman?
我遇到了错误。请帮我解决这个问题?
results <- rfe(x,y, sizes=c(1:13), rfeControl=control , method="svmRadial")
Error in { : task 1 failed – "missing value where TRUE/FALSE needed"
此外:有 50 个或更多警告(使用 warnings() 查看前 50 个)
听到这个消息很遗憾,也许这些提示会有帮助
https://machinelearning.org.cn/faq/single-faq/why-does-the-code-in-the-tutorial-not-work-for-me
感谢您的宝贵信息。我正在使用 ANN、RF、SVM、KNN、NB 和 XGB 进行预测分析。我使用 caret 包为 SVM、KNN 和 NB 计算特征重要性,而对于 ANN、RF 和 XGB,我分别使用了 neuralnetwork、ranomforest 和 xgboost 包。我的问题是,这些包如何选择对不同参数进行排名,因为每个参数的权重在所有 4 个包中并不相同。谢谢
不客气。
我认为每个模型都使用了默认的超参数。如果时间允许,我建议您调整每个算法。
rfe 函数绘图的结果是 RMSE,你是如何得出准确率图的?
用于创建这些图表的代码在上面的教程中。
嗨,Jason,
我想告诉你,我使用了 caret 包的支持向量回归模型。对于我的项目,我还需要考虑自变量的重要性。
你能告诉我,caret 包中的 ‘VarImp’ 函数是如何用于 SVR 等没有内置重要性得分的模型来确定自变量重要性的吗?
祝好,
Younes
你好 Younes… 这里有一些很好的例子
https://topepo.github.io/caret/variable-importance.html
你好 James,感谢如此清晰的教程!
我很难在我的数据上实现这一点,我的数据混合了数值和独热编码的数据。当我只使用数值数据时,一切正常,但当我加入独热编码数据(即 1 和 0)时,我会收到“Error: wrong model type for regression”(错误:回归的模型类型错误)。你知道如何解决这个问题吗?
非常感谢!
你好 Jen… 不客气!你可能会对以下讨论感兴趣
https://stackoverflow.com/questions/23357855/wrong-model-type-for-regression-error-in-10-fold-cross-validation-for-naive-baye
https://www.reddit.com/r/statistics/comments/8q35w7/lasso_regression_caret_error_wrong_model_type_for/