多类别分类问题是指需要预测一个标签,但该标签可能存在两个以上的选项。
这些是具有挑战性的预测建模问题,因为模型需要足够有代表性的每个类别的示例才能学习问题。当每个类别的示例数量不平衡,或者偏向于少数几个类别而其他类别示例很少时,问题会变得更具挑战性。
这类问题被称为不平衡多类别分类问题,它们需要仔细设计评估指标、测试框架以及选择机器学习模型。E.coli 蛋白质定位位点数据集是用于探索不平衡多类别分类挑战的标准数据集。
在本教程中,您将了解如何为不平衡多类别的 E.coli 数据集开发和评估模型。
完成本教程后,您将了解:
- 如何加载和探索数据集,并为数据准备和模型选择提供思路。
- 如何使用稳健的测试框架系统地评估一套机器学习模型。
- 如何拟合最终模型并使用它来预测特定示例的类别标签。
立即开始您的项目,阅读我的新书 《Python 不平衡分类》,其中包含分步教程和所有示例的Python 源代码文件。
让我们开始吧。
- 2021 年 1 月更新:更新了 API 文档链接。

使用大肠杆菌数据集进行不平衡多类别分类
照片来自 Marcus,部分权利保留。
教程概述
本教程分为五个部分;它们是:
- E.coli 数据集
- 探索数据集
- 模型测试和基线结果
- 评估模型
- 评估机器学习算法
- 评估数据过采样
- 在新数据上进行预测
E.coli 数据集
在此项目中,我们将使用一个标准的不平衡机器学习数据集,称为“E.coli”数据集,也称为“蛋白质定位位点”数据集。
该数据集描述了使用 E.coli 蛋白质的氨基酸序列在其细胞定位位点进行分类的问题。也就是说,根据蛋白质折叠前的化学成分来预测蛋白质如何与细胞结合。
该数据集的功劳归于 Kenta Nakai,并由 Paul Horton 和 Kenta Nakai 在他们 1996 年的论文《用于预测蛋白质细胞定位位点的概率分类系统》中发展成现在的形式。在其中,他们取得了 81% 的分类准确率。
336 个 E.coli 蛋白质被分为 8 类,准确率为 81%……
— 《用于预测蛋白质细胞定位位点的概率分类系统》,1996 年。
该数据集包含 336 个 E.coli 蛋白质示例,每个示例都使用从蛋白质氨基酸序列计算出的七个输入变量来描述。
忽略序列名称,输入特征描述如下:
- mcg:McGeoch 的信号序列识别方法。
- gvh:von Heijne 的信号序列识别方法。
- lip:von Heijne 的信号肽 II 共有序列评分。
- chg:预测的脂蛋白 N-末端的电荷存在性。
- aac:外膜和周质蛋白氨基酸含量判别分析得分。
- alm1:ALOM 跨膜区域预测程序的得分。
- alm2:ALOM 程序在排除序列中的推测切割信号区域后的得分。
有八类,描述如下:
- cp:细胞质
- im:无信号序列的内膜
- pp:周质
- imU:内膜,不可切割信号序列
- om:外膜
- omL:外膜脂蛋白
- imL:内膜脂蛋白
- imS:内膜,可切割信号序列
类别之间的示例分布不均,在某些情况下甚至严重不平衡。
例如,“cp”类别有 143 个示例,而“imL”和“imS”类别每个只有两个示例。
接下来,我们仔细看看数据。
想要开始学习不平衡分类吗?
立即参加我为期7天的免费电子邮件速成课程(附示例代码)。
点击注册,同时获得该课程的免费PDF电子书版本。
探索数据集
首先,下载并解压缩数据集,将其保存在当前工作目录中,文件名为“ecoli.csv”。
请注意,此版本的数据集已删除第一列(序列名称),因为它不包含可泛化的建模信息。
查看文件内容。
文件的前几行应如下所示
|
1 2 3 4 5 6 |
0.49,0.29,0.48,0.50,0.56,0.24,0.35,cp 0.07,0.40,0.48,0.50,0.54,0.35,0.44,cp 0.56,0.40,0.48,0.50,0.49,0.37,0.46,cp 0.59,0.49,0.48,0.50,0.52,0.45,0.36,cp 0.23,0.32,0.48,0.50,0.55,0.25,0.35,cp ... |
我们可以看到,输入变量都是数值型,类别标签是字符串值,在建模之前需要进行标签编码。
可以使用 read_csv() Pandas 函数将数据集加载为 DataFrame,指定文件位置并说明没有标题行。
|
1 2 3 4 5 |
... # 定义数据集位置 filename = 'ecoli.csv' # 将csv文件加载为数据框 dataframe = read_csv(filename, header=None) |
加载后,我们可以通过打印DataFrame的形状来总结行数和列数。
|
1 2 3 |
... # 总结数据集的形状 print(dataframe.shape) |
接下来,我们可以计算每个输入变量的五数概括。
|
1 2 3 4 |
... # 描述数据集 set_option('precision', 3) print(dataframe.describe()) |
最后,我们还可以使用 Counter 对象来汇总每个类别的示例数量。
|
1 2 3 4 5 6 7 |
... # 总结类别分布 target = dataframe.values[:,-1] counter = Counter(target) for k,v in counter.items(): per = v / len(target) * 100 print('Class=%s, Count=%d, Percentage=%.3f%%' % (k, v, per)) |
总而言之,下面列出了加载和汇总数据集的完整示例。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 |
# 加载并汇总数据集 from pandas import read_csv from pandas import set_option from collections import Counter # 定义数据集位置 filename = 'ecoli.csv' # 将csv文件加载为数据框 dataframe = read_csv(filename, header=None) # 总结数据集的形状 print(dataframe.shape) # 描述数据集 set_option('precision', 3) print(dataframe.describe()) # 总结类别分布 target = dataframe.values[:,-1] counter = Counter(target) for k,v in counter.items(): per = v / len(target) * 100 print('Class=%s, Count=%d, Percentage=%.3f%%' % (k, v, per)) |
运行示例首先加载数据集,并确认行数和列数,为 336 行,7 个输入变量和 1 个目标变量。
回顾每个变量的摘要,似乎变量已经过中心化,即移动以使均值为 0.5。变量似乎也已归一化,意味着所有值都在 0 到 1 之间;至少没有变量的值超出此范围。
然后汇总类别分布,证实了每个类别观察值的严重倾斜。我们可以看到,“cp”类别占主导地位,占示例的约 42%,而“imS”、“imL”和“omL”等少数类别仅占数据集的 1% 或更少。
这些少数类别可能没有足够的数据进行泛化。一种方法可能是简单地删除这些类别的示例。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 |
(336, 8) 0 1 2 3 4 5 6 count 336.000 336.000 336.000 336.000 336.000 336.000 336.000 mean 0.500 0.500 0.495 0.501 0.500 0.500 0.500 std 0.195 0.148 0.088 0.027 0.122 0.216 0.209 min 0.000 0.160 0.480 0.500 0.000 0.030 0.000 25% 0.340 0.400 0.480 0.500 0.420 0.330 0.350 50% 0.500 0.470 0.480 0.500 0.495 0.455 0.430 75% 0.662 0.570 0.480 0.500 0.570 0.710 0.710 max 0.890 1.000 1.000 1.000 0.880 1.000 0.990 Class=cp, Count=143, Percentage=42.560% Class=im, Count=77, Percentage=22.917% Class=imS, Count=2, Percentage=0.595% Class=imL, Count=2, Percentage=0.595% Class=imU, Count=35, Percentage=10.417% Class=om, Count=20, Percentage=5.952% Class=omL, Count=5, Percentage=1.488% Class=pp, Count=52, Percentage=15.476% |
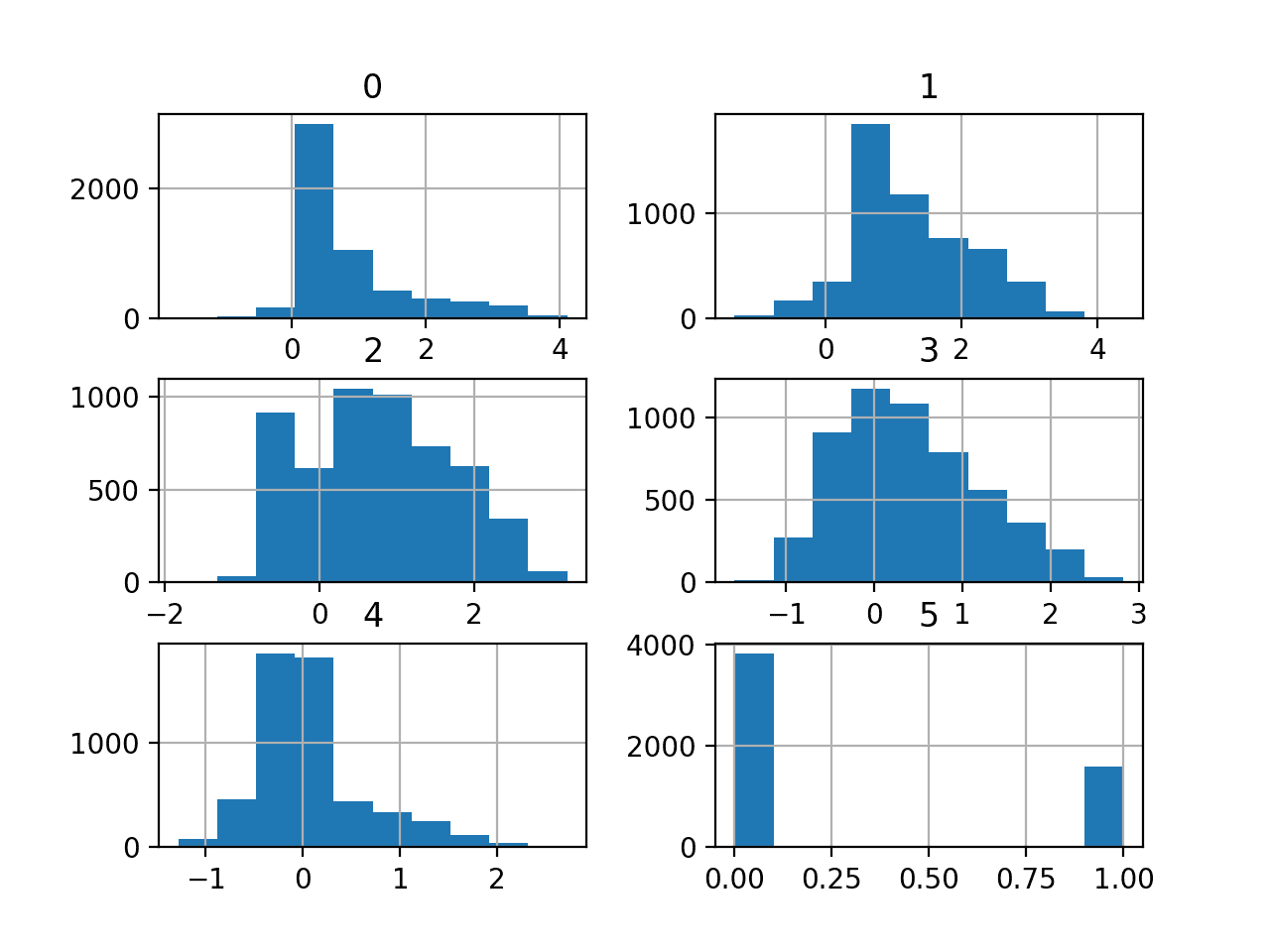
我们还可以通过为每个变量创建直方图来查看输入变量的分布。
下面列出了创建所有输入变量直方图的完整示例。
|
1 2 3 4 5 6 7 8 9 10 11 |
# 创建所有变量的直方图 from pandas import read_csv from matplotlib import pyplot # 定义数据集位置 filename = 'ecoli.csv' # 将csv文件加载为数据框 df = read_csv(filename, header=None) # 创建每个变量的直方图 df.hist(bins=25) # 显示绘图 pyplot.show() |
我们可以看到,像 0、5 和 6 这样的变量可能具有多峰分布。变量 2 和 3 可能具有二元分布,而变量 1 和 4 可能具有 类高斯分布。
根据模型的选择,数据集可能需要标准化、归一化,甚至幂变换。
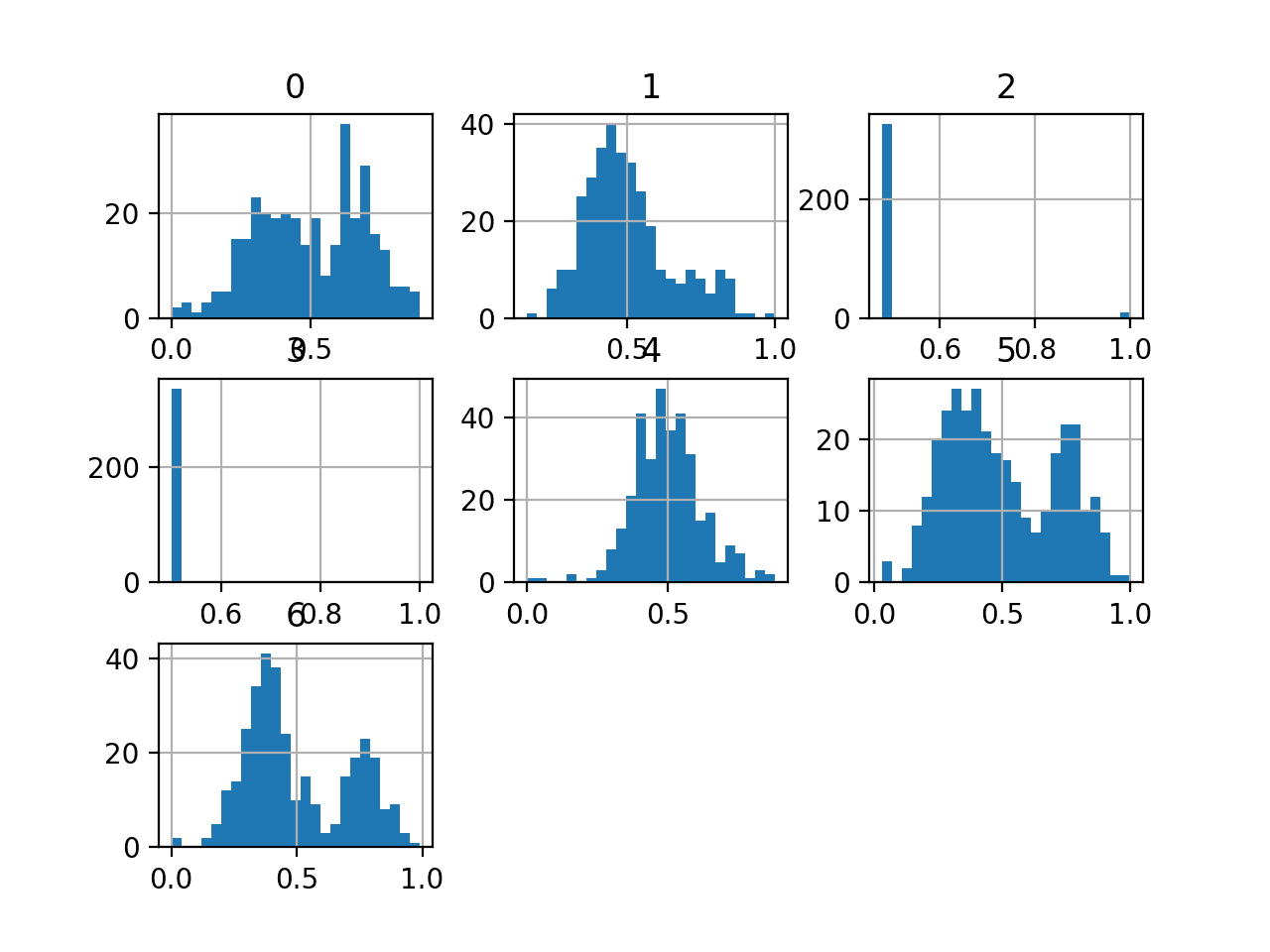
E.coli 数据集变量的直方图
现在我们已经审阅了数据集,接下来我们将开发一个测试工具来评估候选模型。
模型测试和基线结果
k 折交叉验证程序提供了对模型性能的良好通用估计,至少与单次训练-测试划分相比,它不太可能过于乐观地产生偏差。我们将使用k=5,这意味着每折包含约 336/5 或约 67 个示例。
分层表示每折都将尝试包含与整个训练数据集相同的类别示例混合。重复表示将执行多次评估过程,以帮助避免偶然结果并更好地捕捉所选模型的方差。我们将使用三次重复。
这意味着将对单个模型进行 5 * 3 = 15 次拟合和评估,并报告这些运行的平均值和标准差。
这可以使用 scikit-learn 的 RepeatedStratifiedKFold 类来实现。
所有类别都同样重要。因此,在这种情况下,我们将使用分类准确率来评估模型。
首先,我们可以定义一个函数来加载数据集,将输入变量分割为输入和输出变量,并使用标签编码器确保类别标签按顺序编号。
|
1 2 3 4 5 6 7 8 9 10 11 |
# 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 data = read_csv(full_path, header=None) # 检索numpy数组 data = data.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码,使其具有类别0和1 y = LabelEncoder().fit_transform(y) return X, y |
我们可以定义一个函数来使用分层重复 5 折交叉验证来评估候选模型,然后返回模型在每次折叠和重复中计算出的得分列表。
下面的evaluate_model()函数实现了这一点。
|
1 2 3 4 5 6 7 |
# 评估模型 def evaluate_model(X, y, model): # 定义评估过程 cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1) # 评估模型 scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1) return scores |
然后我们可以调用load_dataset()函数来加载并确认 E.coli 数据集。
|
1 2 3 4 5 6 7 |
... # 定义数据集位置 full_path = 'ecoli.csv' # 加载数据集 X, y = load_dataset(full_path) # 总结已加载的数据集 print(X.shape, y.shape, Counter(y)) |
在这种情况下,我们将评估在所有情况下都预测多数类别的基线策略。
这可以使用DummyClassifier类并通过将“strategy”设置为“most_frequent”来自动实现,该类将预测训练数据集中最常见的类别(例如,类别‘cp’)。因此,考虑到训练数据集中最常见类别的分布,我们预计此模型将获得约 42% 的分类准确率。
|
1 2 3 |
... # 定义参考模型 model = DummyClassifier(strategy='most_frequent') |
然后,我们可以通过调用我们的 evaluate_model() 函数来评估模型,并报告结果的平均值和标准差。
|
1 2 3 4 5 |
... # 评估模型 scores = evaluate_model(X, y, model) # 总结性能 print('Mean Accuracy: %.3f (%.3f)' % (mean(scores), std(scores))) |
将所有这些内容结合起来,下面列出了在 E.coli 数据集上评估基线模型的完整示例。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 |
# E.coli 数据集的基线模型和测试框架 from collections import Counter from numpy import mean from numpy import std from pandas import read_csv from sklearn.preprocessing import LabelEncoder from sklearn.model_selection import cross_val_score from sklearn.model_selection import RepeatedStratifiedKFold from sklearn.dummy import DummyClassifier # 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 data = read_csv(full_path, header=None) # 检索numpy数组 data = data.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码,使其具有类别0和1 y = LabelEncoder().fit_transform(y) 返回 X, y # 评估模型 def evaluate_model(X, y, model): # 定义评估过程 cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1) # 评估模型 scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1) 返回 分数 # 定义数据集位置 full_path = 'ecoli.csv' # 加载数据集 X, y = load_dataset(full_path) # 总结已加载的数据集 print(X.shape, y.shape, Counter(y)) # 定义参考模型 model = DummyClassifier(strategy='most_frequent') # 评估模型 scores = evaluate_model(X, y, model) # 总结性能 print('Mean Accuracy: %.3f (%.3f)' % (mean(scores), std(scores))) |
运行示例首先加载数据集,并如预期的那样将案例数量报告为 336,并将类别标签分布报告出来。
然后使用重复分层 k 折交叉验证评估具有默认策略的DummyClassifier,报告的分类准确率平均值和标准差约为 42.6%。
|
1 2 |
(336, 7) (336,) Counter({0: 143, 1: 77, 7: 52, 4: 35, 5: 20, 6: 5, 3: 2, 2: 2}) Mean Accuracy: 0.426 (0.006) |
在模型评估过程中会报告警告;例如
|
1 |
警告:y 中人口最少的类别只有 2 个成员,这太少了。任何类别的最小成员数不能少于 n_splits=5。 |
这是因为某些类别没有足够的示例进行 5 折交叉验证,例如“imS”和“imL”类别。
在这种情况下,我们将从数据集中删除这些示例。这可以通过更新load_dataset()来删除具有这些类别的行来实现,例如四行。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
# 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 df = read_csv(full_path, header=None) # 删除少数类别的行 df = df[df[7] != 'imS'] df = df[df[7] != 'imL'] # 检索numpy数组 data = df.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码,使其具有类别0和1 y = LabelEncoder().fit_transform(y) return X, y |
然后我们可以重新运行示例以建立分类准确率的基线。
完整的示例如下所示。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 |
# E.coli 数据集的基线模型和测试框架 from collections import Counter from numpy import mean from numpy import std from pandas import read_csv from sklearn.preprocessing import LabelEncoder from sklearn.model_selection import cross_val_score from sklearn.model_selection import RepeatedStratifiedKFold from sklearn.dummy import DummyClassifier # 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 df = read_csv(full_path, header=None) # 删除少数类别的行 df = df[df[7] != 'imS'] df = df[df[7] != 'imL'] # 检索numpy数组 data = df.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码,使其具有类别0和1 y = LabelEncoder().fit_transform(y) 返回 X, y # 评估模型 def evaluate_model(X, y, model): # 定义评估过程 cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1) # 评估模型 scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1) 返回 分数 # 定义数据集位置 full_path = 'ecoli.csv' # 加载数据集 X, y = load_dataset(full_path) # 总结已加载的数据集 print(X.shape, y.shape, Counter(y)) # 定义参考模型 model = DummyClassifier(strategy='most_frequent') # 评估模型 scores = evaluate_model(X, y, model) # 总结性能 print('Mean Accuracy: %.3f (%.3f)' % (mean(scores), std(scores))) |
运行示例确认示例数量从 336 减少到 332。
我们还可以看到类别数量从八个减少到六个(类别 0 到类别 5)。
性能基线已确定为 43.1%。此分数提供了此数据集的基线,所有其他分类算法都可以与之比较。达到高于约 43.1% 的分数表示模型在此数据集上具有技能,而达到此值或更低的分数表示模型在此数据集上没有技能。
|
1 2 |
(332, 7) (332,) Counter({0: 143, 1: 77, 5: 52, 2: 35, 3: 20, 4: 5}) Mean Accuracy: 0.431 (0.005) |
现在我们有了测试工具和性能基线,我们可以开始评估该数据集上的一些模型。
评估模型
在本节中,我们将使用上一节中开发的测试工具,评估数据集上的一系列不同技术。
报告的性能良好,但尚未高度优化(例如,超参数未进行调整)。
你能做得更好吗?如果你能使用相同的测试框架获得更好的分类准确率,我很想听听。请在下面的评论中告诉我。
评估机器学习算法
让我们开始在数据集上评估一系列机器学习模型。
在数据集上快速检查一套不同的非线性算法是一个好主意,这样可以快速找出哪些有效并值得进一步关注,哪些无效。
我们将评估以下机器学习模型在 E.coli 数据集上的表现:
- 线性判别分析 (LDA)
- 支持向量机 (SVM)
- 装袋决策树(BAG)
- 随机森林 (RF)
- 极端随机树(ET)
我们将使用大多数默认模型超参数,除了集成算法中的树的数量,我们将将其设置为合理的默认值1000。
我们将依次定义每个模型并将它们添加到一个列表中,以便我们可以按顺序评估它们。下面的 get_models() 函数定义了要评估的模型列表,以及用于稍后绘制结果的模型简称列表。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 |
# 定义要测试的模型 定义 获取_模型(): models, names = list(), list() # LDA models.append(LinearDiscriminantAnalysis()) names.append('LDA') # SVM models.append(LinearSVC()) names.append('SVM') # Bagging models.append(BaggingClassifier(n_estimators=1000)) names.append('BAG') # RF models.append(RandomForestClassifier(n_estimators=1000)) names.append('RF') # ET models.append(ExtraTreesClassifier(n_estimators=1000)) names.append('ET') return models, names |
然后我们可以依次枚举模型列表并评估每个模型,存储分数以供后续评估。
|
1 2 3 4 5 6 7 8 9 10 11 |
... # 定义模型 models, names = get_models() results = list() # 评估每个模型 for i in range(len(models)): # 评估模型并存储结果 scores = evaluate_model(X, y, models[i]) results.append(scores) # 总结性能 print('>%s %.3f (%.3f)' % (names[i], mean(scores), std(scores))) |
在运行结束时,我们可以将每个样本的分数绘制成箱须图,并具有相同的比例,以便直接比较其分布。
|
1 2 3 4 |
... # 绘制结果图 pyplot.boxplot(results, labels=names, showmeans=True) pyplot.show() |
将所有这些内容结合起来,下面列出了在 E.coli 数据集上评估一系列机器学习算法的完整示例。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 |
# 在 E.coli 数据集上快速检查机器学习算法 from numpy import mean from numpy import std from pandas import read_csv from matplotlib import pyplot from sklearn.preprocessing import LabelEncoder from sklearn.model_selection import cross_val_score from sklearn.model_selection import RepeatedStratifiedKFold 从 sklearn.svm 导入 LinearSVC from sklearn.discriminant_analysis import LinearDiscriminantAnalysis from sklearn.ensemble import RandomForestClassifier from sklearn.ensemble import ExtraTreesClassifier from sklearn.ensemble import BaggingClassifier # 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 df = read_csv(full_path, header=None) # 删除少数类别的行 df = df[df[7] != 'imS'] df = df[df[7] != 'imL'] # 检索numpy数组 data = df.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码 y = LabelEncoder().fit_transform(y) 返回 X, y # 评估模型 def evaluate_model(X, y, model): # 定义评估过程 cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1) # 评估模型 scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1) 返回 分数 # 定义要测试的模型 定义 获取_模型(): models, names = list(), list() # LDA models.append(LinearDiscriminantAnalysis()) names.append('LDA') # SVM models.append(LinearSVC()) names.append('SVM') # Bagging models.append(BaggingClassifier(n_estimators=1000)) names.append('BAG') # RF models.append(RandomForestClassifier(n_estimators=1000)) names.append('RF') # ET models.append(ExtraTreesClassifier(n_estimators=1000)) names.append('ET') return models, names # 定义数据集位置 full_path = 'ecoli.csv' # 加载数据集 X, y = load_dataset(full_path) # 定义模型 models, names = get_models() results = list() # 评估每个模型 for i in range(len(models)): # 评估模型并存储结果 scores = evaluate_model(X, y, models[i]) results.append(scores) # 总结性能 print('>%s %.3f (%.3f)' % (names[i], mean(scores), std(scores))) # 绘制结果图 pyplot.boxplot(results, labels=names, showmeans=True) pyplot.show() |
运行该示例将依次评估每个算法,并报告分类准确率的平均值和标准差。
注意:由于算法或评估程序的随机性,或数值精度的差异,您的结果可能会有所不同。请考虑运行示例几次并比较平均结果。
在这种情况下,我们可以看到所有测试的算法都具有技能,其准确率均高于默认的 43.1%。
结果表明,大多数算法在此数据集上表现良好,并且可能是树决策的集成算法表现最好,其中 Extra Trees 达到 88% 的准确率,Random Forest 达到 89.5% 的准确率。
|
1 2 3 4 5 |
>LDA 0.886 (0.027) >SVM 0.883 (0.027) >BAG 0.851 (0.037) >RF 0.895 (0.032) >ET 0.880 (0.030) |
会创建一个图表,显示每个算法样本结果的一个箱线图。箱体显示数据中间的 50%,每个箱体中间的橙色线显示样本的中位数,每个箱体中的绿色三角形显示样本的平均值。
我们可以看到,树决策集成算法的得分分布聚集在一起,与其他测试的算法分开。在大多数情况下,均值和中位数在图中很接近,这表明得分分布具有一定程度的对称性,可能表明模型是稳定的。
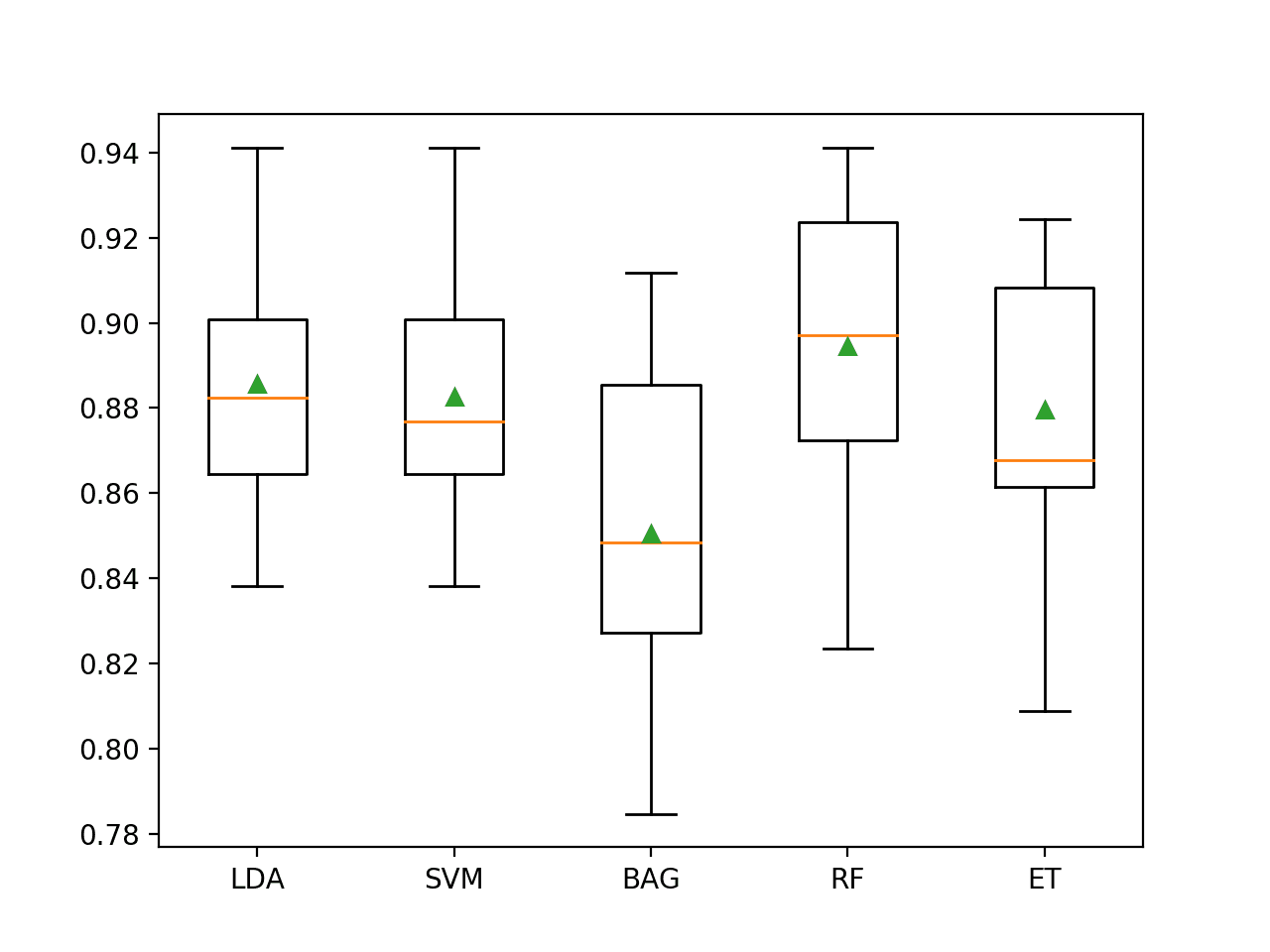
在不平衡的 E.coli 数据集上机器学习模型的箱线图
评估数据过采样
由于类别众多,许多类别中的示例很少,该数据集可能受益于过采样。
我们可以测试 SMOTE 算法应用于除多数类别(cp)之外的所有类别,这可以提高性能。
通常,SMOTE 对树决策集成似乎没有帮助,因此我们将测试的算法集更改为以下内容:
- 多项逻辑回归 (LR)
- 线性判别分析 (LDA)
- 支持向量机 (SVM)
- k-近邻(KNN)
- 高斯过程 (GP)
下面列出了更新的get_models()函数版本,用于定义这些模型。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 |
# 定义要测试的模型 定义 获取_模型(): models, names = list(), list() # LR models.append(LogisticRegression(solver='lbfgs', multi_class='multinomial')) names.append('LR') # LDA models.append(LinearDiscriminantAnalysis()) names.append('LDA') # SVM models.append(LinearSVC()) names.append('SVM') # KNN models.append(KNeighborsClassifier(n_neighbors=3)) names.append('KNN') # GP models.append(GaussianProcessClassifier()) names.append('GP') return models, names |
我们可以使用 imbalanced-learn 库中的 SMOTE 实现,以及来自同一库的 Pipeline,首先将 SMOTE 应用于训练数据集,然后在交叉验证过程中拟合给定的模型。
SMOTE 将使用训练数据集中的 k-近邻来合成新示例,默认情况下,k 设置为 5。
这对于我们数据集中的一些类别来说太大了。因此,我们将尝试将k值设为 2。
|
1 2 3 4 5 6 |
... # 创建管道 steps = [('o', SMOTE(k_neighbors=2)), ('m', models[i])] pipeline = Pipeline(steps=steps) # 评估模型并存储结果 scores = evaluate_model(X, y, pipeline) |
将这一切结合起来,下面列出了在 E.coli 数据集上使用 SMOTE 过采样处理的完整示例。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 |
# 在 E.coli 数据集上使用 SMOTE 与机器学习算法进行快速检查 from numpy import mean from numpy import std from pandas import read_csv from matplotlib import pyplot from sklearn.preprocessing import LabelEncoder from sklearn.model_selection import cross_val_score from sklearn.model_selection import RepeatedStratifiedKFold 从 sklearn.svm 导入 LinearSVC from sklearn.discriminant_analysis import LinearDiscriminantAnalysis from sklearn.neighbors import KNeighborsClassifier from sklearn.gaussian_process import GaussianProcessClassifier from sklearn.linear_model import LogisticRegression from imblearn.pipeline import Pipeline from imblearn.over_sampling import SMOTE # 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 df = read_csv(full_path, header=None) # 删除少数类别的行 df = df[df[7] != 'imS'] df = df[df[7] != 'imL'] # 检索numpy数组 data = df.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码 y = LabelEncoder().fit_transform(y) 返回 X, y # 评估模型 def evaluate_model(X, y, model): # 定义评估过程 cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1) # 评估模型 scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1) 返回 分数 # 定义要测试的模型 定义 获取_模型(): models, names = list(), list() # LR models.append(LogisticRegression(solver='lbfgs', multi_class='multinomial')) names.append('LR') # LDA models.append(LinearDiscriminantAnalysis()) names.append('LDA') # SVM models.append(LinearSVC()) names.append('SVM') # KNN models.append(KNeighborsClassifier(n_neighbors=3)) names.append('KNN') # GP models.append(GaussianProcessClassifier()) names.append('GP') return models, names # 定义数据集位置 full_path = 'ecoli.csv' # 加载数据集 X, y = load_dataset(full_path) # 定义模型 models, names = get_models() results = list() # 评估每个模型 for i in range(len(models)): # 创建管道 steps = [('o', SMOTE(k_neighbors=2)), ('m', models[i])] pipeline = Pipeline(steps=steps) # 评估模型并存储结果 scores = evaluate_model(X, y, pipeline) results.append(scores) # 总结性能 print('>%s %.3f (%.3f)' % (names[i], mean(scores), std(scores))) # 绘制结果图 pyplot.boxplot(results, labels=names, showmeans=True) pyplot.show() |
运行该示例将依次评估每个算法,并报告分类准确率的平均值和标准差。
注意:由于算法或评估程序的随机性,或数值精度的差异,您的结果可能会有所不同。请考虑运行示例几次并比较平均结果。
在这种情况下,我们可以看到,SMOTE 带来的 LDA 准确率从 88.6% 下降到约 87.9%,而 SVM 结合 SMOTE 的准确率则从约 88.3% 小幅提高到约 88.8%。
在这种情况下,SVM 似乎也是最佳表现方法,尽管它没有像前一节中的随机森林那样取得改进。
|
1 2 3 4 5 |
>LR 0.875 (0.024) >LDA 0.879 (0.029) >SVM 0.888 (0.025) >KNN 0.835 (0.040) >GP 0.876 (0.023) |
创建每个算法分类准确率分数的箱线图。
我们可以看到 LDA 在 90% 以上的性能上有许多异常值,这非常有趣。这可能表明,如果 LDA 专注于数量丰富的类别,它的表现可能会更好。
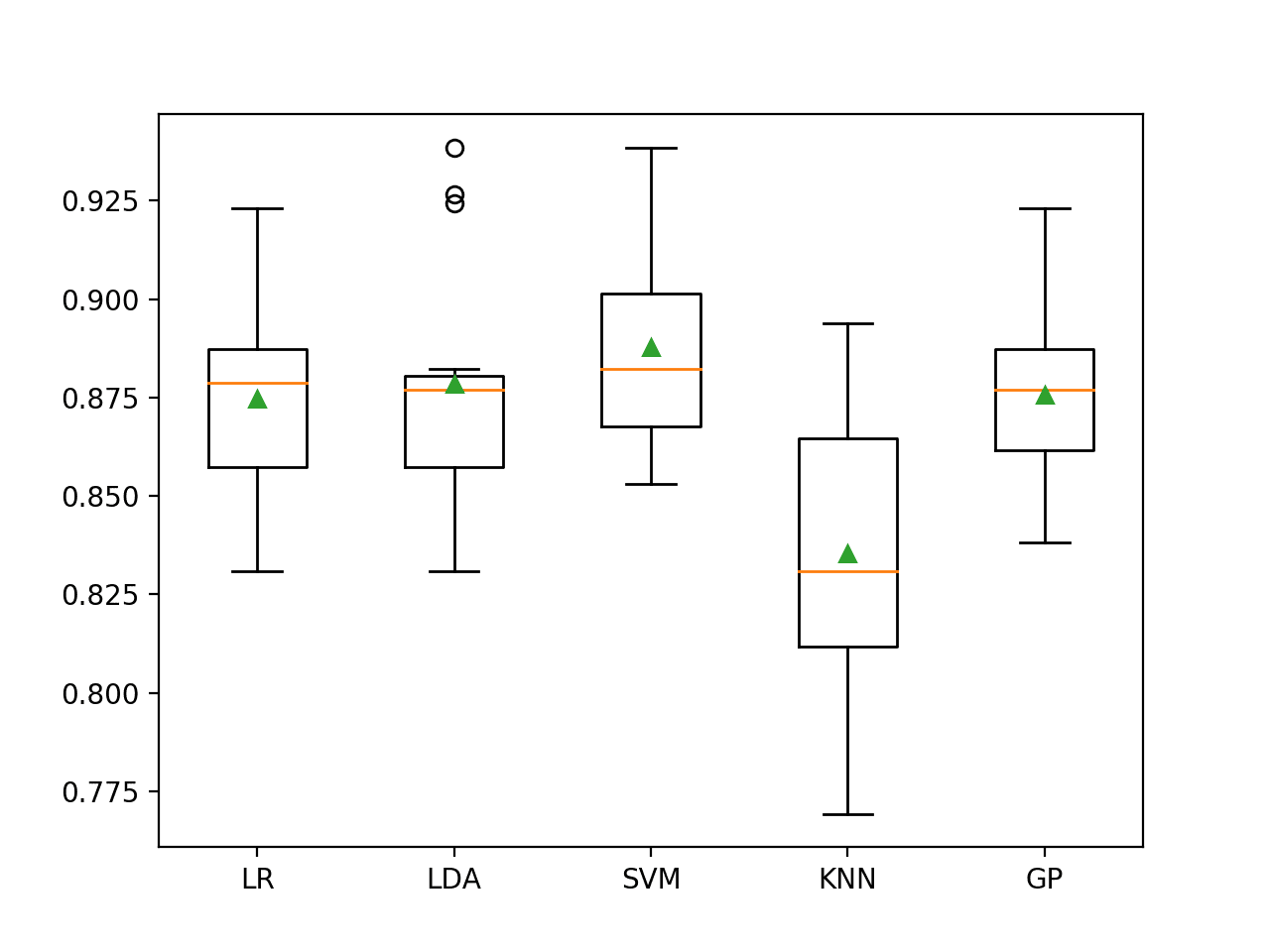
SMOTE 与机器学习模型在不平衡的 E.coli 数据集上的箱线图
现在我们已经了解了如何在此数据集上评估模型,接下来看看如何使用最终模型进行预测。
在新数据上进行预测
在本节中,我们可以拟合最终模型并使用它来预测单行数据。
我们将使用随机森林模型作为我们的最终模型,该模型实现了约 89.5% 的分类准确率。
首先,我们可以定义模型。
|
1 2 3 |
... # 定义要评估的模型 model = RandomForestClassifier(n_estimators=1000) |
定义好后,我们就可以在整个训练数据集上对其进行拟合。
|
1 2 3 |
... # 拟合模型 model.fit(X, y) |
拟合后,我们可以通过调用 predict() 函数来使用它为新数据进行预测。这将返回每个示例的编码类别标签。
然后,我们可以使用标签编码器进行反向转换,以获得字符串类别标签。
例如
|
1 2 3 4 5 6 |
... # 定义一行数据 row = [...] # 预测类别标签 yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] |
为了演示这一点,我们可以使用已训练好的模型来预测一些已知结果的案例的标签。
完整的示例如下所示。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 |
# 在 ecoli 数据集上拟合模型并进行预测 from pandas import read_csv from sklearn.preprocessing import LabelEncoder from sklearn.ensemble import RandomForestClassifier # 加载数据集 def load_dataset(full_path): # 将数据集加载为numpy数组 df = read_csv(full_path, header=None) # 删除少数类别的行 df = df[df[7] != 'imS'] df = df[df[7] != 'imL'] # 检索numpy数组 data = df.values # 分割为输入和输出元素 X, y = data[:, :-1], data[:, -1] # 对目标变量进行标签编码 le = LabelEncoder() y = le.fit_transform(y) return X, y, le # 定义数据集位置 full_path = 'ecoli.csv' # 加载数据集 X, y, le = load_dataset(full_path) # 定义要评估的模型 model = RandomForestClassifier(n_estimators=1000) # 拟合模型 model.fit(X, y) # 已知类别 "cp" row = [0.49,0.29,0.48,0.50,0.56,0.24,0.35] yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] print('>Predicted=%s (expected cp)' % (label)) # 已知类别 "im" row = [0.06,0.61,0.48,0.50,0.49,0.92,0.37] yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] print('>Predicted=%s (expected im)' % (label)) # 已知类别 "imU" row = [0.72,0.42,0.48,0.50,0.65,0.77,0.79] yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] print('>Predicted=%s (expected imU)' % (label)) # 已知类别 "om" row = [0.78,0.68,0.48,0.50,0.83,0.40,0.29] yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] print('>Predicted=%s (expected om)' % (label)) # 已知类别 "omL" row = [0.77,0.57,1.00,0.50,0.37,0.54,0.0] yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] print('>Predicted=%s (expected omL)' % (label)) # 已知类别 "pp" row = [0.74,0.49,0.48,0.50,0.42,0.54,0.36] yhat = model.predict([row]) label = le.inverse_transform(yhat)[0] print('>Predicted=%s (expected pp)' % (label)) |
首先运行示例,在整个训练数据集上拟合模型。
然后,将拟合后的模型用于预测来自六个类别中每个类别的示例的标签。
我们可以看到,为每个选定的示例都正确预测了类别标签。尽管如此,平均而言,我们预计 10 次预测中会有 1 次是错误的,而这些错误在不同类别之间可能不均等。
|
1 2 3 4 5 6 |
>Predicted=cp (expected cp) >Predicted=im (expected im) >Predicted=imU (expected imU) >Predicted=om (expected om) >Predicted=omL (expected omL) >Predicted=pp (expected pp) |
进一步阅读
如果您想深入了解,本节提供了更多关于该主题的资源。
论文
- 革兰氏阴性菌蛋白质定位位点预测专家系统, 1991.
- 真核细胞蛋白质定位位点预测的知识库, 1992.
- 用于预测蛋白质细胞定位位点的概率分类系统, 1996.
API
- pandas.read_csv API.
- sklearn.dummy.DummyClassifier API.
- imblearn.over_sampling.SMOTE API.
- imblearn.pipeline.Pipeline API.
数据集 (Dataset)
总结
在本教程中,您将学习如何为不平衡的多类别 E.coli 数据集开发和评估模型。
具体来说,你学到了:
- 如何加载和探索数据集,并为数据准备和模型选择提供思路。
- 如何使用稳健的测试框架系统地评估一套机器学习模型。
- 如何拟合最终模型并使用它来预测特定示例的类别标签。
你有什么问题吗?
在下面的评论中提出你的问题,我会尽力回答。




好的,Jason!很棒的代码!
我有几个问题,希望您能 kindly 回答。
SVM 和 KNN 分类器的性能比随机森林分类器更好,速度快约 17 倍,准确率只差 1%,为什么不使用这些算法而不是随机森林来进行最终预测呢?我想这些性能(执行时间)在处理海量数据时会产生巨大的影响。
您能否在您的博客中包含一个教程,例如“如何在 KFold 验证中使用管道:优缺点?管道与 KFolding 从零开始?”
是否有其他关于 E.coli 的数据集可以运行先前训练好的模型来预测未知目标?
干杯!!!
也许可以。
模型选择应基于项目需求,例如,不仅考虑模型性能,还要考虑方差和计算复杂度。
https://machinelearning.org.cn/a-gentle-introduction-to-model-selection-for-machine-learning/
当然。您对管道具体有什么疑问?也许可以给我发邮件。
https://machinelearning.org.cn/contact/
抱歉,我不明白您最后一个问题。也许您可以详细说明一下?
你好 Jason,
我能否从您提供的分类示例中受益于功能强大的 Nvidia GPU,如 GTX 2080?
此致
Dom
否,示例在 CPU 上运行。
哈喽 杰森,
首先,感谢您通过您的杰出工作邀请我们进入这个精彩的世界!
我想请您评论一下这个结果(10000 个训练向量,每个向量是 60 个估计器)
使用您的 Python 代码和 evaluate_model 在 ExtraTreesClassifier 上
我得到的准确率是 83.3%
而在验证(即在训练数据上使用 model.fit 函数,在未见过的数据上使用 model.predict)时,我的准确率是 53%。
我该如何解释这些结果?
模型是否没有泛化到从未见过的数据?
提前感谢
Dom
模型在训练集上表现良好,在测试集上表现不佳。
它没有很好地泛化,或者测试数据集不具代表性。
嗨,Jason,
感谢您的回复……
所以模型没有泛化……好的……这很清楚……
但我不明白的是
为什么使用 evaluate_model 在 ExtraTreesClassifier 上可以得到 83.3% 的准确率
而在测试数据上实际准确率是 53%?
我不明白。
在验证期间,我有
训练数据:Train_X,Train_y
model.fit(Train_X,Train_y)
print(‘model.score=’,model.score(Train_X,Train_y)) -> 100%
验证数据:Valid_X,Valid_y
print(‘model.score=’,model.score(Valid_X,Valid_y)) -> 53%
我不明白……我是不是做错了什么?
提前感谢
Dom
通过训练集评估模型是模型性能的无效估计。
这就是为什么我们使用留出集或交叉验证。
在您的情况下,唯一的评估是估计的 val 集合性能,即 53%。
非常感谢分享,Jason!一如既往地富有启发性。
我有一个关于选择“准确率”作为度量标准的问题。考虑到类别不平衡,您会推荐其他分数吗?我知道对于不平衡的二分类问题可以选择 G-mean、balanced accuracy 等,但它们也适用于多分类问题吗?
提前表示感谢!
不客气。
是的,我喜欢这个数据集的准确率。
是的,您可以选择另一个度量标准,请参阅此教程。
https://machinelearning.org.cn/tour-of-evaluation-metrics-for-imbalanced-classification/
您好 Jason!由于过采样没有提高准确率,我们是否可以得出结论,数据不平衡在这里不是一个“问题”?为什么它不是一个问题?是因为我们查看了准确率,但没有关注每个类别的召回率和精确率(或标准化混淆矩阵的对角线)吗?是因为特征足够预测目标,所以我们能够做得比基线准确率好得多吗?
是的。您需要查看召回率、精确率以及特征与结果的关系。考虑一些夸张的例子:如果失衡是十亿比一,即使我过采样了一百次,如果我总是预测出现次数最多的结果,我仍然能获得相同的准确率。如果结果是与特征无关的随机数,那么无论使用什么技术,模型都无法提高。想想您的准确率是过高还是过低。然后您就可以大致了解是什么问题了。
以下代码片段在 y 中返回一个值为 ‘0’,从而导致类似以下的错误:“ValueError: y 中最不常见的类只有一个成员,太少了。任何类的最小组数不能少于 2。”
X, y = data[:, :-1], data[:, -1]
# 对目标变量进行标签编码,使其具有类别 0 和 1
y = LabelEncoder().fit_transform(y)
您好 MaMo……感谢提问。
我很想帮忙,但我实在没有能力为您调试代码。
我很乐意提出一些建议
考虑将代码积极削减到最低要求。这将帮助您隔离问题并专注于它。
考虑将问题简化为一个或几个简单的例子。
考虑寻找其他可行的类似代码示例,并慢慢修改它们以满足您的需求。这可能会暴露您的失误。
考虑在 StackOverflow 上发布您的问题和代码。