为你的问题找到一个好的机器学习算法很难。但一旦找到了,你如何从中获得最佳性能呢?
在这篇文章中,你将发现三种在R中调整机器学习算法参数的方法。
通过一个真实示例,一步一步地学习R中的工作代码。将代码用作模板,在你当前或下一个机器学习项目上调整机器学习算法。
通过我的新书《R语言机器学习精通》,开始你的项目,其中包含分步教程和所有示例的R源代码文件。
让我们开始吧。

在R中调整随机森林。
照片由Susanne Nilsson拍摄,保留部分权利。
从顶级算法中获得更好的准确性
为你的数据集找到一个好的甚至表现良好的机器学习算法是很困难的。
通过反复试验的过程,你可以确定一个有潜力的算法短列表,但你如何知道哪个是最好的呢?
你可以为每个算法使用默认参数。这些参数是由经验法则或书籍和研究论文中的建议设定的。但你怎么知道你选择的算法是否展现了它们最佳的性能呢?
使用算法调整来搜索算法参数
答案是为你的问题搜索好的甚至最佳的算法参数组合。
你需要一个流程来调整每个机器学习算法,这样你才知道你从中获得了最大的收益。调整完成后,你就可以对你短列表中的算法进行客观的比较。
搜索算法参数可能很困难,有很多选择,例如
- 要调整哪些参数?
- 使用哪种搜索方法来定位好的算法参数?
- 使用什么测试选项来限制对训练数据的过拟合?
需要更多关于R机器学习的帮助吗?
参加我为期14天的免费电子邮件课程,了解如何在您的项目中使用R(附带示例代码)。
点击注册,同时获得该课程的免费PDF电子书版本。
在R中调整机器学习算法
你可以在R中调整你的机器学习算法参数。
通常,本节中的方法假定你已经为你正在寻找更好性能的问题选择了一个表现良好的机器学习算法短列表。
创建算法短列表的一个绝佳方法是使用caret包。
有关如何使用caret包的更多信息,请参阅
在本节中,我们将介绍三种你可以在R中用于调整算法参数的方法。
- 使用caret R包。
- 使用算法自带的工具。
- 设计你自己的参数搜索。
在开始调整之前,让我们设置好我们的环境和测试数据。
测试设置
让我们快速看一下数据和我们将在本案例研究中使用的算法。
测试数据集
在本案例研究中,我们将使用声纳测试问题。
这是一个来自UCI机器学习数据库的数据集,它描述了雷达回波是撞击金属还是岩石。
这是一个二分类问题,有60个数值输入特征,描述了雷达回波的属性。你可以在这里了解更多关于这个问题的信息:声纳数据集。你可以在这里看到该数据集的世界级发表结果:声纳数据集上的准确率。
这个数据集并非特别困难,但它并非微不足道,对这个例子很有趣。
让我们加载所需的库,并从mlbench包加载数据集。
|
1 2 3 4 5 6 7 8 9 |
library(randomForest) library(mlbench) library(caret) # 加载数据集 data(Sonar) dataset <- Sonar x <- dataset[,1:60] y <- dataset[,61] |
测试算法
我们将使用流行的随机森林算法作为我们算法调整的主题。
随机森林不一定是这个数据集的最佳算法,但它是一个非常流行的算法,毫无疑问,调整它将是你自己机器学习工作中一项有用的练习。
在调整算法时,了解你的算法非常重要,这样你才能知道参数对你正在创建的模型有什么影响。
在本案例研究中,我们将坚持调整两个参数,即mtry和ntree参数,它们对我们的随机森林模型有以下影响。还有许多其他参数,但这两个参数可能是对最终准确度影响最大的参数。
直接来自R中randomForest()函数的帮助页面
- mtry:每次拆分时随机采样的变量数量。
- ntree:要生成的树的数量。
让我们通过使用每个参数的推荐默认值和mtry=floor(sqrt(ncol(x)))或mtry=7和ntree=500来创建一个基线进行比较。
|
1 2 3 4 5 6 7 8 9 |
# 使用默认参数创建模型 control <- trainControl(method="repeatedcv", number=10, repeats=3) seed <- 7 metric <- "Accuracy" set.seed(seed) mtry <- sqrt(ncol(x)) tunegrid <- expand.grid(.mtry=mtry) rf_default <- train(Class~., data=dataset, method="rf", metric=metric, tuneGrid=tunegrid, trControl=control) print(rf_default) |
我们可以看到估计的准确率为81.3%。
|
1 2 3 4 |
重采样结果 准确率 Kappa 准确率SD Kappa SD 0.8138384 0.6209924 0.0747572 0.1569159 |
1. 使用Caret进行调整
R中的caret包为调整机器学习算法参数提供了出色的功能。
caret中并非所有机器学习算法都可用。参数的选择留给包的开发者,即Max Khun。只有那些影响很大(例如,在Khun看来确实需要调整)的算法参数才能在caret中进行调整。
因此,只有mtry参数在caret中可用。原因是它对最终准确率的影响,并且它必须针对数据集进行经验查找。
ntree参数不同,因为它可以任意大,并且持续增加准确率直到某个点。它的调整难度较小,也非关键,并且可能更多地受可用计算时间限制,而不是其他任何因素。
随机搜索
我们可以使用的一种搜索策略是在范围内尝试随机值。
如果我们不确定值可能是多少,并且我们想克服我们在设置参数时可能存在的任何偏见(例如上述建议的方程),这可能很有用。
让我们尝试使用caret进行mtry的随机搜索。
|
1 2 3 4 5 6 7 |
# 随机搜索 control <- trainControl(method="repeatedcv", number=10, repeats=3, search="random") set.seed(seed) mtry <- sqrt(ncol(x)) rf_random <- train(Class~., data=dataset, method="rf", metric=metric, tuneLength=15, trControl=control) print(rf_random) plot(rf_random) |
请注意,我们正在使用一个类似于用于检查算法的测试框架。10折交叉验证和3次重复都会减慢搜索过程,但目的是限制和减少训练集上的过拟合。它不会完全消除过拟合。如果能留出验证集进行最终检查,那将是一个很好的主意。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
跨调优参数的重采样结果 mtry 准确率 Kappa 准确率SD Kappa SD 11 0.8218470 0.6365181 0.09124610 0.1906693 14 0.8140620 0.6215867 0.08475785 0.1750848 17 0.8030231 0.5990734 0.09595988 0.1986971 24 0.8042929 0.6002362 0.09847815 0.2053314 30 0.7933333 0.5798250 0.09110171 0.1879681 34 0.8015873 0.5970248 0.07931664 0.1621170 45 0.7932612 0.5796828 0.09195386 0.1887363 47 0.7903896 0.5738230 0.10325010 0.2123314 49 0.7867532 0.5673879 0.09256912 0.1899197 50 0.7775397 0.5483207 0.10118502 0.2063198 60 0.7790476 0.5513705 0.09810647 0.2005012 |
我们可以看到,mtry最准确的值是11,准确率为82.1%。
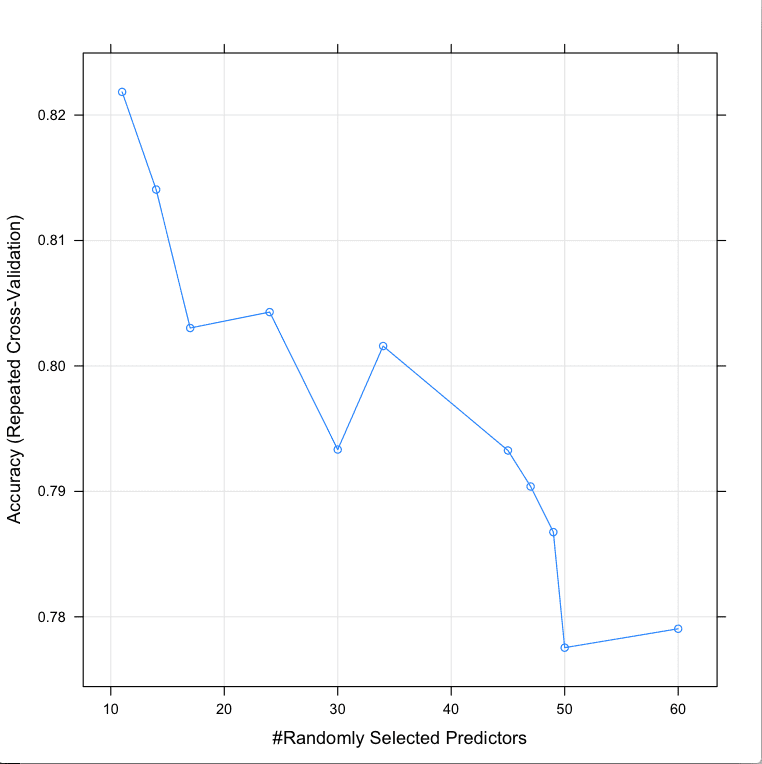
在R中使用随机搜索调整随机森林参数
网格搜索
另一种搜索方法是定义一个要尝试的算法参数网格。
网格的每个轴都是一个算法参数,网格中的点是特定的参数组合。由于我们只调整一个参数,网格搜索是通过候选值向量进行的线性搜索。
|
1 2 3 4 5 6 |
control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid") set.seed(seed) tunegrid <- expand.grid(.mtry=c(1:15)) rf_gridsearch <- train(Class~., data=dataset, method="rf", metric=metric, tuneGrid=tunegrid, trControl=control) print(rf_gridsearch) plot(rf_gridsearch) |
我们可以看到,mtry最准确的值是2,准确率为83.78%。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 |
跨调优参数的重采样结果 mtry 准确率 Kappa 准确率SD Kappa SD 1 0.8377273 0.6688712 0.07154794 0.1507990 2 0.8378932 0.6693593 0.07185686 0.1513988 3 0.8314502 0.6564856 0.08191277 0.1700197 4 0.8249567 0.6435956 0.07653933 0.1590840 5 0.8268470 0.6472114 0.06787878 0.1418983 6 0.8298701 0.6537667 0.07968069 0.1654484 7 0.8282035 0.6493708 0.07492042 0.1584772 8 0.8232828 0.6396484 0.07468091 0.1571185 9 0.8268398 0.6476575 0.07355522 0.1529670 10 0.8204906 0.6346991 0.08499469 0.1756645 11 0.8073304 0.6071477 0.09882638 0.2055589 12 0.8184488 0.6299098 0.09038264 0.1884499 13 0.8093795 0.6119327 0.08788302 0.1821910 14 0.8186797 0.6304113 0.08178957 0.1715189 15 0.8168615 0.6265481 0.10074984 0.2091663 |
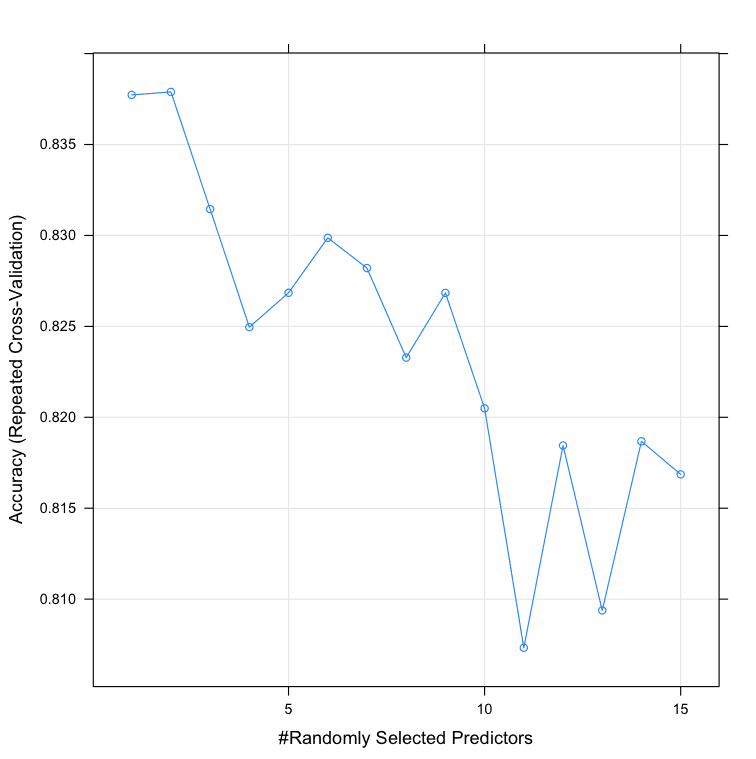
在R中使用网格搜索调整随机森林参数.png
2. 使用算法工具进行调整
一些算法提供了调整算法参数的工具。
例如,randomForest包中的随机森林算法实现提供了tuneRF()函数,该函数会根据你的数据搜索最佳mtry值。
|
1 2 3 4 |
# 算法调整 (tuneRF) set.seed(seed) bestmtry <- tuneRF(x, y, stepFactor=1.5, improve=1e-5, ntree=500) print(bestmtry) |
您可以看到,mtry最准确的值是10,OOBError为0.1442308。
这与我们在上面的caret重复交叉验证实验中看到的不太匹配,其中mtry=10的准确率为82.04%。不过,这是调整算法的另一种方法。
|
1 2 3 4 5 |
mtry OOBError 5.OOB 5 0.1538462 7.OOB 7 0.1538462 10.OOB 10 0.1442308 15.OOB 15 0.1682692 |
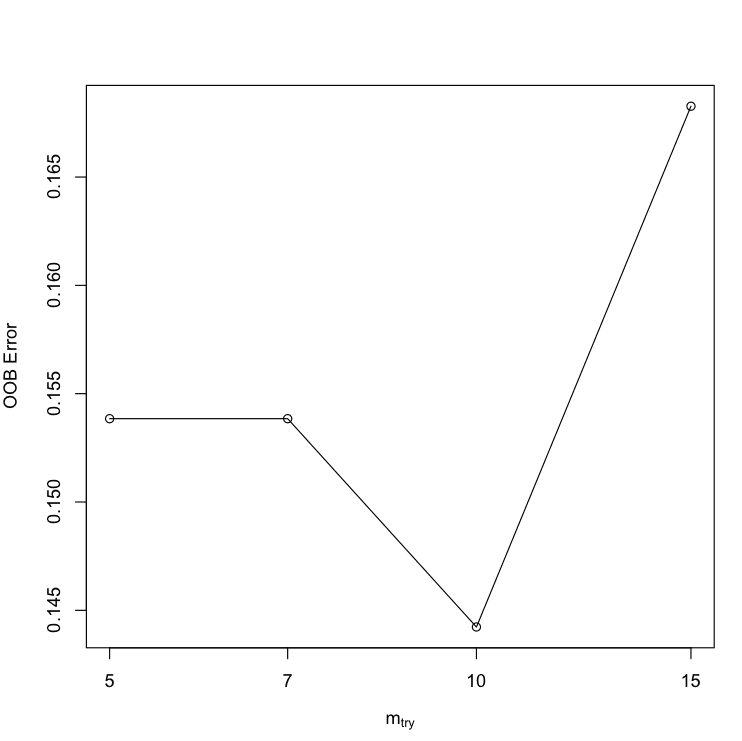
使用tuneRF在R中调整随机森林参数
3. 设计你自己的参数搜索
通常,您想搜索必须调整的参数(由caret处理)以及需要为您的数据集进行缩放或更通用调整的参数。
您必须自己设计参数搜索。
我推荐的两个流行选项是
- 手动调整:编写R代码创建大量模型并使用caret比较它们的准确率。
- 扩展Caret:创建一个caret的扩展,为你想调整的算法添加caret的额外参数。
手动调整
我们希望继续使用caret,因为它提供了一个与我们先前模型直接比较的点(相同的数据分割),并且因为它提供的重复交叉验证测试框架,我们喜欢它,因为它减少了过拟合的严重性。
一种方法是为我们的算法创建许多caret模型,并将不同的参数直接传递给算法。让我们看一个例子,在保持mtry恒定的情况下评估ntree的不同值。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
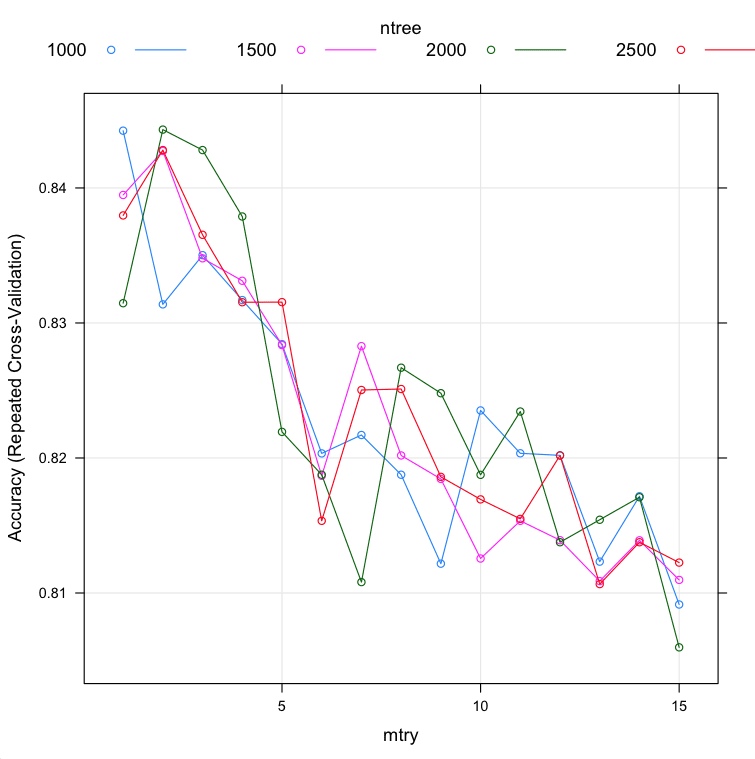
# 手动搜索 control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid") tunegrid <- expand.grid(.mtry=c(sqrt(ncol(x)))) modellist <- list() for (ntree in c(1000, 1500, 2000, 2500)) { set.seed(seed) fit <- train(Class~., data=dataset, method="rf", metric=metric, tuneGrid=tunegrid, trControl=control, ntree=ntree) key <- toString(ntree) modellist[[key]] <- fit } # 比较结果 results <- resamples(modellist) summary(results) dotplot(results) |
可以看到,ntree最准确的值可能是2000,平均准确率为82.02%(比我们第一次使用默认mtry值进行实验的准确率有所提升)。
结果可能表明ntree的最佳值在2000到2500之间。另外请注意,我们将mtry固定为默认值。我们可以重复实验,使用可能更好的mtry=2(来自上述实验),或者尝试ntree和mtry的组合,以防它们有交互作用。
|
1 2 3 4 5 6 7 8 9 |
模型:1000, 1500, 2000, 2500 重采样次数:30 准确度 Min. 1st Qu. Median Mean 3rd Qu. Max. NA's 1000 0.600 0.8024 0.8500 0.8186 0.8571 0.9048 0 1500 0.600 0.8024 0.8095 0.8169 0.8571 0.9500 0 2000 0.619 0.8024 0.8095 0.8202 0.8620 0.9048 0 2500 0.619 0.8000 0.8095 0.8201 0.8893 0.9091 0 |
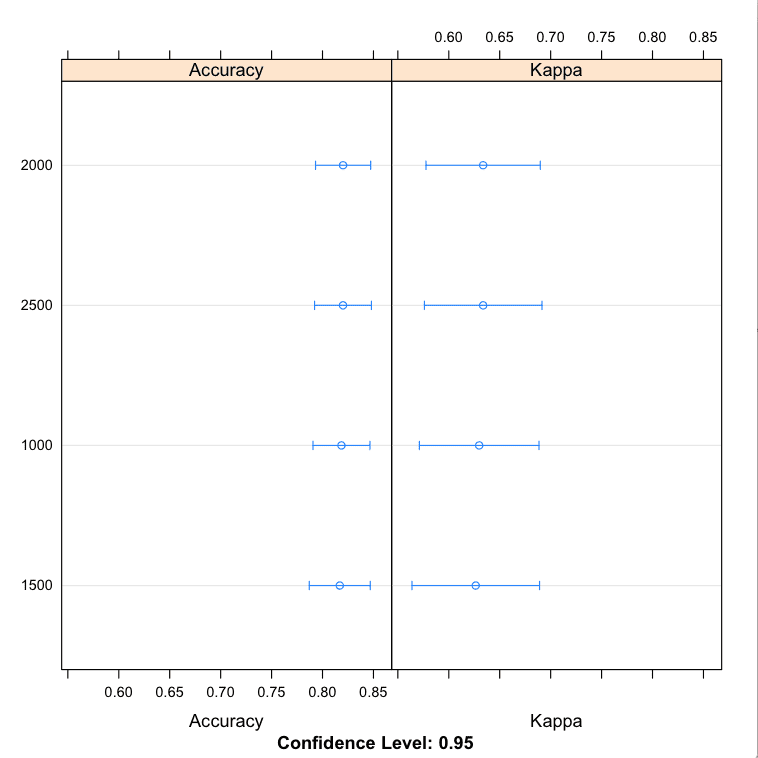
手动在R中调整随机森林参数
扩展Caret
另一种方法是创建一个“新”算法供caret支持。
这与您正在使用的随机森林算法相同,只是进行了修改,使其支持对多个参数进行多次调整。
这种方法的一个风险是,caret对该算法的原生支持包含了额外或花哨的代码包装,这些代码微妙但重要地改变了它的行为。您可能需要用您的自定义算法支持重复之前的实验。
通过定义一个列表,其中包含caret包查找的许多自定义命名元素(例如如何拟合和如何预测),我们可以定义自己的算法以在caret中使用。下面是一个用于caret的自定义随机森林算法的定义,该算法同时接受mtry和ntree参数。
|
1 2 3 4 5 6 7 8 9 10 11 12 |
customRF <- list(type = "Classification", library = "randomForest", loop = NULL) customRF$parameters <- data.frame(parameter = c("mtry", "ntree"), class = rep("numeric", 2), label = c("mtry", "ntree")) customRF$grid <- function(x, y, len = NULL, search = "grid") {} customRF$fit <- function(x, y, wts, param, lev, last, weights, classProbs, ...) { randomForest(x, y, mtry = param$mtry, ntree=param$ntree, ...) } customRF$predict <- function(modelFit, newdata, preProc = NULL, submodels = NULL) predict(modelFit, newdata) customRF$prob <- function(modelFit, newdata, preProc = NULL, submodels = NULL) predict(modelFit, newdata, type = "prob") customRF$sort <- function(x) x[order(x[,1]),] customRF$levels <- function(x) x$classes |
现在,让我们在caret train函数调用中使用这个自定义列表,并尝试调整ntree和mtry的不同值。
|
1 2 3 4 5 6 7 |
# 训练模型 control <- trainControl(method="repeatedcv", number=10, repeats=3) tunegrid <- expand.grid(.mtry=c(1:15), .ntree=c(1000, 1500, 2000, 2500)) set.seed(seed) custom <- train(Class~., data=dataset, method=customRF, metric=metric, tuneGrid=tunegrid, trControl=control) summary(custom) plot(custom) |
这可能需要一两分钟才能运行。
您可以看到,ntree和mtry最准确的值分别是2000和2,准确率为84.43%。
我们确实看到ntree的数量和ntree的值之间可能存在一些交互作用。尽管如此,如果我们选择了通过网格搜索找到的最佳mtry值2(上面)和通过网格搜索找到的最佳ntree值2000(上面),在这种情况下,我们将获得与此组合搜索相同的调整水平。这是一个很好的确认。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 |
mtry ntree 准确率 Kappa 准确率SD Kappa SD 1 1000 0.8442424 0.6828299 0.06505226 0.1352640 1 1500 0.8394805 0.6730868 0.05797828 0.1215990 1 2000 0.8314646 0.6564643 0.06630279 0.1381197 1 2500 0.8379654 0.6693773 0.06576468 0.1375408 2 1000 0.8313781 0.6562819 0.06909608 0.1436961 2 1500 0.8427345 0.6793793 0.07005975 0.1451269 2 2000 0.8443218 0.6830115 0.06754346 0.1403497 2 2500 0.8428066 0.6791639 0.06488132 0.1361329 3 1000 0.8350216 0.6637523 0.06530816 0.1362839 3 1500 0.8347908 0.6633405 0.06836512 0.1418106 3 2000 0.8428066 0.6800703 0.06643838 0.1382763 3 2500 0.8365296 0.6668480 0.06401429 0.1336583 4 1000 0.8316955 0.6574476 0.06292132 0.1317857 4 1500 0.8331241 0.6605244 0.07543919 0.1563171 4 2000 0.8378860 0.6699428 0.07147459 0.1488322 4 2500 0.8315368 0.6568128 0.06981259 0.1450390 5 1000 0.8284343 0.6505097 0.07278539 0.1516109 5 1500 0.8283622 0.6506604 0.07166975 0.1488037 5 2000 0.8219336 0.6375155 0.07548501 0.1564718 5 2500 0.8315440 0.6570792 0.07067743 0.1472716 6 1000 0.8203391 0.6341073 0.08076304 0.1689558 6 1500 0.8186797 0.6302188 0.07559694 0.1588256 6 2000 0.8187590 0.6310555 0.07081621 0.1468780 6 2500 0.8153463 0.6230495 0.07728249 0.1623253 7 1000 0.8217027 0.6367189 0.07649651 0.1606837 7 1500 0.8282828 0.6503808 0.06628953 0.1381925 7 2000 0.8108081 0.6147563 0.07605609 0.1573067 7 2500 0.8250361 0.6437397 0.07737756 0.1602434 8 1000 0.8187590 0.6314307 0.08378631 0.1722251 8 1500 0.8201876 0.6335679 0.07380001 0.1551340 8 2000 0.8266883 0.6472907 0.06965118 0.1450607 8 2500 0.8251082 0.6434251 0.07745300 0.1628087 9 1000 0.8121717 0.6177751 0.08218598 0.1709987 9 1500 0.8184488 0.6300547 0.08077766 0.1674261 9 2000 0.8247980 0.6429315 0.07260439 0.1513512 9 2500 0.8186003 0.6302674 0.07356916 0.1547231 10 1000 0.8235209 0.6407121 0.07991334 0.1656978 10 1500 0.8125541 0.6183581 0.06851683 0.1421993 10 2000 0.8187518 0.6308120 0.08538951 0.1782368 10 2500 0.8169336 0.6263682 0.07847066 0.1649216 11 1000 0.8203463 0.6341158 0.07222587 0.1497558 11 1500 0.8153463 0.6235878 0.09131621 0.1904418 11 2000 0.8234416 0.6402906 0.07586609 0.1576765 11 2500 0.8154906 0.6236875 0.07485835 0.1576576 12 1000 0.8201948 0.6336913 0.08672139 0.1806589 12 1500 0.8139105 0.6206994 0.08638618 0.1804780 12 2000 0.8137590 0.6204461 0.07771424 0.1629707 12 2500 0.8201876 0.6333194 0.07799832 0.1636237 13 1000 0.8123232 0.6173280 0.09299062 0.1936232 13 1500 0.8108802 0.6142721 0.08416414 0.1760527 13 2000 0.8154257 0.6236191 0.08079923 0.1693634 13 2500 0.8106566 0.6138814 0.08074394 0.1687437 14 1000 0.8171645 0.6270292 0.08608806 0.1799346 14 1500 0.8139033 0.6207263 0.08522205 0.1781396 14 2000 0.8170924 0.6276518 0.08766645 0.1822010 14 2500 0.8137590 0.6207371 0.08353328 0.1746425 15 1000 0.8091486 0.6110154 0.08455439 0.1745129 15 1500 0.8109668 0.6154780 0.08928549 0.1838700 15 2000 0.8059740 0.6047791 0.08829659 0.1837809 15 2500 0.8122511 0.6172771 0.08863418 0.1845635 |
在R中自定义调整随机森林参数
有关在caret中定义自定义算法的更多信息,请参阅
要查看caret使用的随机森林的实际包装器,你可以将其用作起点,请参阅
总结
在这篇文章中,你发现了调整表现良好的机器学习算法以获得最佳性能的重要性。
你完成了一个在R中调整随机森林算法的示例,并发现了调整表现良好的算法的三种方法。
- 使用caret R包。
- 使用算法自带的工具。
- 设计你自己的参数搜索。
你现在拥有了一个工作示例和模板,可以用于在你当前或下一个项目中调整R中的机器学习算法。
下一步
完成本文中的示例。
- 打开你的R交互环境。
- 键入或复制粘贴示例代码。
- 慢慢来,理解发生了什么,使用R帮助文档来阅读函数。
你有什么问题吗?在评论区提问,我会尽力回答。




虽然我尝试使用树的数量和mtry参数来调整随机森林模型,但结果相同。表格看起来像这样,我需要预测y11。
x11 x12 x13 x14 x15 x16 x17 x18 x19 y11
0 0 0 2 0 2 2 4 0.000000000 ?
1 1 0 0 0 3 3 18 0.000025700 ?
0 1 0 0 1 2 2 2 0.000000000 ?
5 2 1 0 1 12 12 14 0.000128479
0 0 2 0 1 3 3 3 0.000000000
4 0 2 0 1 7 8 104 0.000102783
所有x11到x19都是检测y11值的关键特征。我这样设置了模型,但仍然无法达到高效率。你能帮帮我吗?
rf_model <- randomForest(y11~x11+x12+x13+x14+x15+x16+x17+x18+x19, data =new_dataframe_train,ntree=1001,importance=TRUE,keep.forest=TRUE,mtry=3)
rf_pred<-predict(rf_model,new_dataframe_test)
通常,调整mtry并持续增加树的数量直到性能不再有提升是一个好主意。
随机森林无需进行特征选择,它会自动进行,忽略那些分割点不好的特征。
安装包时出错
包“videoplayR”在 R 版本 3.5.2 中不可用
我不知道那个包,抱歉。或许可以试试发布到 Stack Overflow 或 R 用户组?
嗨,Jason,
感谢您的帖子。我一直在尝试在嵌套交叉验证的 rf 后获得顶部预测变量的稳定性,就像我在 glmnet 中那样,但我遇到了困难。您有什么建议吗?
谢谢!
Emma
抱歉,您说的“预测变量上的稳定性”是什么意思?
感谢您的代码。我正在尝试运行它们,但我无法获得与您在最后一部分中相同的输出
> summary(custom)
> plot(custom)
对于“summary(custom)”,我得到类似这样的结果:
长度 类 模式
call 5 -none- call
type 1 -none- character
predicted 10537 factor numeric
err.rate 7500 -none- numeric
confusion 6 -none- numeric
votes 21074 matrix numeric
oob.times 10537 -none- numeric
classes 2 -none- character
importance 51 -none- numeric
importanceSD 0 -none- NULL
localImportance 0 -none- NULL
proximity 0 -none- NULL
ntree 1 -none- numeric
mtry 1 -none- numeric
forest 14 -none- list
y 10537 factor numeric
test 0 -none- NULL
inbag 0 -none- NULL
xNames 51 -none- character
problemType 1 -none- character
tuneValue 2 data.frame list
obsLevels 2 -none- character
你能告诉我哪里出错了什么吗?代码运行了 4 天,我对此感到失望,因为我没有得到预期的输出。谢谢!
试试
> custom$results.
嗨,Jason,
我爱你的网站和工作。
在运行随机森林算法之前,通常会对数据进行标准化并使用 PCA 减少特征数量吗?我理解 RF 在拆分节点时会选择最佳特征,但我使用 PCA 的唯一动机是缩短计算时间。
Aditya,谢谢。
不,我建议让 RF 自己来处理。
如果有时间,你可以稍后比较一下 PCA 投影版本的数据集,看看它是否能超越原始数据。
太好了。谢谢您的提示!
很棒的工作!感谢您的帖子!🙂
回归的情况下,指标是什么?
Harini,谢谢,很高兴您觉得它有用。
当您写模型的那一行时
rf_default <- train(Class~., . . . )
我的 R 编译器说“Class”未找到,Class~. 代表什么?一个人如何解决这个问题?
这是公式表示法,其中“Class”是数据集中变量的名称。它可能不存在于您的数据集中。
关于公式表示法,请参见此处:
https://stat.ethz.ch/R-manual/R-devel/library/stats/html/formula.html
和这里
https://ww2.coastal.edu/kingw/statistics/R-tutorials/formulae.html
非常有信息量的博客!谢谢!我正在尝试自定义 RF 函数用于回归模型。我更改了 type = “regression” 和 metric=”RMSE”。但是,对于 custom <- train(x,y, method=customRF, metric="RMSE", tuneGrid=tunegrid, trControl=c – 我收到错误。“错误在 train.default(x, y, method = customRF, metric = "RMSE", tuneGrid = tunegrid,
回归模型类型错误”。有什么可能出错了?
同时,我按照下面的代码操作了:
control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid")
tunegrid <- expand.grid(.mtry=c(1:15))
modellist <- list()
for (ntree in c(1000, 1500, 2000, 2500,3000)) {
fit <- train(x,y, method="rf", metric="RMSE", tuneGrid=tunegrid, trControl=control, ntree=ntree)
key <- toString(ntree)
modellist[[key]] <- fit
}
但是这花费的时间太长了!
奇怪,看起来它不想将随机森林用于回归。
试试在你的数据集上不带网格搜索地使用 caret train() 的“rf”,并确保它能工作。
Jason,谢谢,我按照你的建议试了一下,fit$predicted 的输出是 NULL!
在函数帮助中,提到“train{caret} 函数为许多分类和回归例程设置了调优参数的网格,拟合每个模型并计算基于重采样的方法的性能度量。”
鉴于不进行网格搜索的情况,我上面代码中获得的调优参数是否相关?
你可以调用 predict() 函数来进行预测。
mtry 是否有一个理论上的上限?我看到大多数示例中的上限是 15,但为什么不是例如 35 呢?任何参考都将非常感谢:)
嗨 DF,
mtry 是每次拆分时考虑的特征数量。
可能的取值范围是从 1 到数据集中特征的数量。
嗨,Jason,
感谢您的帖子,非常详细且超级清晰!为了跟进 DF 的问题:我理解 mtry 的可能取值范围是从 1 到特征的数量。这是否意味着如果我想对 mtry 进行系统性调优,如果我有大量特征(例如超过 5,000 个),我应该尝试所有这些值?选择大的 mtry 值有什么优缺点?任何参考都将不胜感激!
不,通常 1 到 sqrt(特征数量) 就足够了。
调优它的影响取决于具体问题。
这里有一个在 Python 中调优它的例子
https://machinelearning.org.cn/random-forest-ensemble-in-python/
Jason,感谢您的回复!但我发现如果我选择大于 sqrt(特征数量) 的值,模型性能(准确率)会更好。我可以使用更大的 mtry 值吗?还是必须严格使用 < sqrt(特征数量)?这个 sqrt(特征数量) 的原理是什么?
太好了。是的,你可以。
这只是一个启发式方法。
亲爱的 Jason,
正如您所说,mtry 的上限是数据样本中属性的数量。我不明白这一点。
您可以看到(如下所示)我的 trainData 中有 11 个属性。我复制了您的代码,其中 mtry 设置为 mtry <- sqrt(ncol(trainData))。为什么结果显示模型尝试了许多 mtry 值?最好的 mtry = 25!这比数据样本的属性还要多。
control <- trainControl(method="repeatedcv", number=10, repeats=3, search="random")
set.seed(2222)
mtry <- sqrt(ncol(trainData))
rf_random ncol(trainData)
[1] 12
结果
随机森林
623 个样本
11 个预测变量
2 个类别:“0”,“1”
无预处理
重采样:交叉验证(10 折,重复 3 次)
样本量汇总:561、560、561、560、561、561、……
跨调优参数的重采样结果
mtry Accuracy Kappa
1 0.7122839 0.3097868
4 0.8138063 0.5970567
10 0.8149683 0.6041117
16 0.8128004 0.5994635
17 0.8106752 0.5951853
22 0.8149336 0.6048981
24 0.8149939 0.6053784
25 0.8160003 0.6069581
31 0.8122969 0.5985465
35 0.8016210 0.5760874
37 0.8037374 0.5809098
38 0.8053330 0.5842086
准确度用于使用最大值选择最优模型。
用于模型的最终值为 mtry = 25。
我无法解释这一点,这非常奇怪!
也许您的场景中存在错误的假设?或者您的代码有错误?
嗨,Jason,
为了提高模型的准确性,我回去寻找错误分类的训练点并进行纠正或删除。我有数千个训练点,所以很难在训练数据集中找到它们。我想知道,由于 R 中随机森林包的混淆矩阵显示了每个类别中错误分类的训练点数量,是否有办法轻松地精确定位那些错误分类的训练点?我拥有我的训练样本的 ID,但我不知道是否可能找到它们。
删除错误分类点的有趣策略。这通常不是我使用或推荐的方法。
你为什么要这样调整你的模型?
我的一些训练点是使用谷歌地球收集的,所以你可以想象它们并不总是最好的验证数据。这就是我调整训练点的原因。你能给我一些关于这个问题的建议吗?通常可以手动定位这些点,但这很耗时。我确信一定有办法显示错误分类的点,但我不知道如何。
要识别错误分类的点需要另一个信息来源。
这听起来像是在建模之前的、与机器学习算法的选择或使用无关的数据清理练习。
是的,我说的就是“数据清理”,但不是在使用随机森林模型之前。我将未清理的数据用作预分析并获得混淆矩阵。混淆矩阵显示了多少点被错误分类,但我需要确切知道哪些点被错误分类,以便我可以清理它。我需要一个 R 代码来显示错误分类的点。我有每个点的顺序号(总共 3000 个),如果这有帮助的话。
我建议逐一比较预测值和实际点,以找出哪些点被错误分类。
我手头没有示例,抱歉。
感谢您提供的好信息!
但我有一个问题。
>control set.seed(seed)
>tunegrid rf_gridsearch control set.seed(seed)
>mtry rf_random <- train(Class~., data=dataset, method="rf", metric=metric, tuneLength=15, trControl=control)
祝您有美好的一天!!
什么问题?
哈哈哈!
我试图提供帮助……但有时很难。
感谢您提供的好信息。但是,我想了解更多参数,比如 nodesize。那么……如何编写 R 代码呢?🙁
问题被删除了……可能存在错误……
所以我现在申请。
在这个网格搜索代码中,您使用了 set.seed(seed)。同样,您在随机搜索代码中使用了相同的函数。我知道 set.seed 是什么。但我不知道为什么您在网格搜索代码中使用此函数。我知道如果我的信息是错误的,请解释 set.seed 函数在此代码中的作用。祝您有美好的一天!
# 训练模型
control <- trainControl(method="repeatedcv", number=10, repeats=3)
tunegrid <- expand.grid(.mtry=c(1:15), .ntree=c(1000, 1500, 2000, 2500))
set.seed(seed)
custom <- train(Class~., data=dataset, method=customRF, metric=metric, tuneGrid=tunegrid, trControl=control)
summary(custom)
plot(custom)
我调用 seed 来初始化随机数生成器,以便每次运行代码时都能获得相同的结果。这对于教程很有用。
您可以在此处了解更多关于应用机器学习的随机性质
https://machinelearning.org.cn/randomness-in-machine-learning/
哦。感谢您的回复。
起初,我以为 set.seed 函数会影响随机搜索中 ntree 和 mtry 的随机数选择。
但您是说 seed 函数会影响 trainControl 的结果和 train 函数的结果,对吗?
正确。包括拟合模型在内的整个算法评估过程。
嗨 Jason。您介绍了两种很好的方法(网格搜索、随机搜索)。
还有其他方法可以选择最佳参数吗??
我想知道,即使我不设置参数范围,我也能找到模型的最佳参数。随机搜索最终使用 tuneLength 参数来设置数量……。
有方法找到我想要的参数吗?
您可以使用穷举搜索。
我们通常没有资源来找到“最佳”参数,并且乐于使用“良好”或“已找到最佳”的参数。
谢谢!
但是……什么是穷举搜索??
它是函数吗??还是像网格搜索那样尝试很多次??
穷举搜索就像网格搜索,其中网格是所有可能的取值。
https://en.wikipedia.org/wiki/Brute-force_search
嗨 Jason。感谢您的工作。我正在尝试运行“扩展 Caret”部分,但无法获得与您相同的输出,特别是这个:
> summary(custom)
> plot(custom)
“summary(custom)”中的某些东西是错误的,几乎与 Bing 之前提到的 Beth 建议的一样,因为我使用了 custom$results. 结果仍然是错误的。
您能否就此提供进一步的建议?谢谢!
你遇到了什么具体问题?
你好,Jason。
我以前从未尝试过 Caret,我假设下面的代码行执行交叉验证。
control <- trainControl(method="repeatedcv", number=10, repeats=3)
我的问题是,为什么需要交叉验证,因为在随机森林的算法哲学中,它已经是交叉验证的了。
我们需要一个方案来评估模型在未见过的数据上的表现。
我们可以使用训练/测试分割或 k 倍交叉验证。
感谢这篇博文,它给了我信心,让我超越 Coursera ML 课程项目的要求,大胆地不仅在一个基本的 Forest 模型中调整 mtry 参数,还调整了一个具有径向基核的支持向量机模型中的 C 参数。甚至还尝试绘制了 mtry 和 ntree 设置对 OOB 误差的影响。
https://rpubs.com/DrFreedy/WeightLiftingExecisePredictiveModel
我现在在 Twitter 上关注您,也邀请您在 LinkedIn 上连接。🙂
干得好!
你好,
我已给出如下代码:
round5_train_CV<-train(SalePrice~.,data=TrainData_split,method="rf",
trControl=trainControl(method="cv",number=5),
prox=TRUE,allowParallel=TRUE)
训练数据集中有 84 个特征。所以,在任何一点考虑的最大特征数为 84。然而,我得到的输出是 mtry 为 145 和 288,这很奇怪(尝试了超过 84 个特征)。我很困惑。请澄清。
mtry RMSE Rsquared
2 47522.34 0.7675486
145 30352.25 0.8574420
288 30593.40 0.8540389
算法正在报告错误。预测模型的错误与特征数量无关(嗯,不直接相关)。
不是问题。只是想感谢您花时间分享这个。它对我帮助很大。
谢谢 Julian。
网格搜索调优方法效果很好。Jason 干得好,将所有内容集中在一页上很有帮助。
很高兴听到这个消息!
嗨,我听说交叉验证对于随机森林来说不是必需的,因为我们可以使用袋外误差。在这个演示中,我们使用带有 control 的 train 函数。你能给我一些关于它的评论吗?非常感谢!
当然,我也读过同样的内容。
我试图在检查所有算法时保持我的测试框架一致,因此使用了 CV。
感谢您的回复!我明白了。我也听说使用 OOB 误差可能会高估准确率。所以从这个意义上说,用 CV 进行调优,而不是使用 'randomForest' 中的 'tuneRF',可能也很有趣。
你好,
我想知道您是否知道如何重写 RF 模型以允许调整截止概率?例如,如果我们有一个类别不平衡的数据集,是否可以编写一个自定义 RF 模型,其中 mtry 固定为 floor(sqrt(predictors)),并且 cutoff 会迭代一系列值,例如 .1,.2,.3… .9,每个类别?所以第一次迭代的截止值可能是 c(.25,.25,.5),第二次可能是 c(.3,.4.3) 等等?唯一的“窍门”是截止概率必须加起来等于 1。
好问题。
我预计会有论文/代码来针对不平衡数据(例如,内置加权采样)进行自定义随机森林。
你好,
我尝试在随机森林中进行网格搜索。
tunegrid_2 <- expand.grid(.mtry=c(1:7), .ntree=c(1000, 1500, 2000, 2500))
set.seet(1234)
RF_fit <- train(class~., data = tables, method = "rf",metric = "ROC",tuneGrid = tunegrid_2, trControl =control)
但是我得到了类似这样的错误:“Error: The tuning parameter grid should have columns mtry”。
非常感谢您的帮助。
我不知道,抱歉。
这里也是同样的错误!
听起来您需要将“mtry”添加到正在调优的超参数列表中。
是的,我也遇到了同样的问题。在搜索了很多信息后,我发现只有 mtry 可以列出并在 train 方法中进行调优。ntrees 不能同时调优。我不知道如何同时调优它们?
嗨 Melody…请澄清您的问题,以便我能理解您想要完成什么,并更好地帮助您。
你好,
谢谢你的代码。我尝试了 caret 中的 customRF 实现,它可以运行,但不会返回模型重采样性能度量。
警告信息
在 nominalTrainWorkflow(x = x, y = y, wts = weights, info = trainInfo,
重采样性能度量中有缺失值。
有什么想法吗?
谢谢。
嗨,Jason,
谢谢你的博客。这非常有帮助。
我正在评估几种方法的性能(线性回归、随机森林、支持向量机、梯度提升、神经网络和 cubist)用于与回归相关的问题。我正在为此使用 caret 包,并且一直在使用 10 折交叉验证方法。为了比较模型的性能,我使用了 R2 和 RMSE 两个指标。还有其他指标可用于比较这些模型吗?除了这两个指标之外,我还被建议使用一些信息准则来评估模型性能。我唯一知道的信息准则是 AIC 和 BIC,但不确定是否可以与 Caret 包一起使用。您有什么建议吗?
我的另一个问题是关于划分数据的理由。我正在处理 250 条记录。我将数据分为 80(训练)和 20(测试),因为我认为 80:20 是最常用的比例。您能推荐哪种划分适用于哪种场景吗?如果您必须回答“为什么将总数据划分为 80:20”,您的回答是什么?有我可以引用的来源(参考文献)吗?我需要向某人解释
我的另一个问题是关于逐步回归与遗传算法在解决多重共线性问题上的区别。我广泛使用了逐步回归,但没有使用遗传算法。您熟悉这些吗?如果熟悉,您能否帮助我比较它们的优缺点?
对这些有什么建议,我将不胜感激。谢谢。
是的,有很多指标可供使用。仔细思考项目的目标,并选择有助于您展示/未展示您正在实现这些目标的指标。
那个划分是可以的,但如果使用 k 折交叉验证来估计模型技能,则可能不需要。
您可以执行敏感性分析,以显示相同样本量统计属性的变化,以支持选定的样本量。可能不会产生任何实际影响,例如,只是纸上谈兵。
是的,我建议测试一套不同的特征选择方法,根据每种方法的结果构建模型,并加倍投入效果最好的方法。如果您有资源,可以尝试所有特征子集。
谢谢 Jason 的回复。您能否提供一个示例来说明如何使用 AIC 或 BIC 或其他信息准则来评估使用 CARET 包构建的回归模型?我被要求这样做,但不知道如何做。
遗传算法也一样。任何示例都有帮助。谢谢。
感谢您的建议。
嗨,Jason,
谢谢你的博客。这非常有帮助!
我想问一下您在 Extend caret item 中用来调整模型的物理配置是什么?您说花费 1-2 分钟,但我等待了最多 2 小时才完成我的过程。我的物理配置是 (core i7-3720QM RAM 8GB)
应该可以的。也许你用完了内存,或者代码没有使用所有核心?
您好,先生!
感谢非常有帮助的博客!
我想问一下 caret 包中的准确率得分是从哪里获得的?混淆矩阵还是来自袋外数据?
然后,我有一些离题的问题。我知道随机森林主要是为了预测。但是,我们能否使随机森林更具可解释性?或者有没有我们可以使用的包来获取随机森林树的样本?希望您能帮助我回答这个问题。
我在这里有更多关于 caret 中指标的信息
https://machinelearning.org.cn/machine-learning-evaluation-metrics-in-r/
总的来说,我们不关心使机器学习模型可解释,我在这里写了关于这个话题的文章。
https://machinelearning.org.cn/faq/single-faq/how-do-i-interpret-the-predictions-from-my-model
嗨,Jason,
一旦我找到了最佳参数,并想在(之前已分离且未用于调优过程的)数据集上验证模型,我能否直接采用调优过程中找到并训练的最佳模型,还是应该再次从头开始训练模型?如果您能提供帮助,我将非常感激。
(感谢您提供这个非常棒的有用网站!)
当然是您的选择。
我会用最佳超参数在所有训练数据上重新拟合模型,并在留出集上进行评估。
好的,非常感谢!
先生,我将随机森林用作分类方法。我在训练数据和测试数据上评估了模型。在训练数据上,我的准确率是 100%。有什么原因会导致这种情况发生?
问题可能太简单了,不需要机器学习。
嗨,Jason,
感谢您关于随机森林参数调优的精彩博文。我正在尝试使用重复交叉验证(10 折,3 次)来测试我的模型。我应该设置多少种子?是 31 吗?请就设置种子数提供建议。
谢谢你
Priya
使用默认的 m_tree,即特征数量的平方根。
嗨 Jason,
我是您作品和博客的忠实粉丝。我有一个问题,
参数调优是否仅适用于随机森林模型?如果我想开发任何逻辑回归模型,是否也可以为逻辑回归模型调优参数?
诚挚地,
Sayam
当然可以!
通常,GridSearch 和 Algorithmic Tuning 会提供不同的结果,不是吗?那么如何区分和选择它们呢?
选择一个平均性能最佳、模型稳定或模型简单的配置。
您好 Jason,在 caret 包中使用 RF 时,可以获得变量重要性图。在常规 RF 中,这是通过 Gini 减少来实现的——caret RF 变量重要性图是否也显示了 gini 减少?caret 包的文档没有明确说明这一点。
我不确定 caret 包是否可以,但我认为随机森林包可以。我手头没有示例。
我很难修改 customRF 函数来处理新数据集。它也是一个来自 UCI 的数据集(https://archive.ics.uci.edu/ml/datasets/heart+Disease)。它有 13 个变量,我想预测的变量是疾病。有关修改您的函数的任何建议。
任何人
抱歉,我没有好的建议,也许可以尝试将您的具体错误发布到 stackoverflow 或 R 用户列表中?
为什么您选择 mtry=c(1:15)?是因为您的数据集中的预测变量在第 1 到第 15 列吗?
是的,这是每次分割点可能选择的随机特征的数量。
您好,当我为 RF 逻辑回归分类运行时,我得到了以下错误。
Error in metric %in% c(“RMSE”, “Rsquared”) : object ‘metric’ not found
很遗憾听到这个消息,我不知道原因,也许可以尝试发布到 stackoverflow?
接下来我该怎么做,在获得调优参数 ntree 和 mtry 之后。
我应该回到我的 RF 模型并使用调优参数重新构建它吗?
仅调优这两个参数可能就足够了。
非常感谢您,先生,这个博客对我帮助很大。但我可以问一个问题吗?
我在“Extend Caret”部分遇到问题,R 给我返回的错误消息如下。
出错了;所有 Accuracy 指标值都丢失了
Accuracy Kappa
Min. : NA Min. : NA
1st Qu.: NA 1st Qu.: NA
Median : NA Median : NA
Mean :NaN Mean :NaN
3rd Qu.: NA 3rd Qu.: NA
Max. : NA Max. : NA
NA's :30 NA's :30
Error: Stopping
In addition: Warning message
在 nominalTrainWorkflow(x = x, y = y, wts = weights, info = trainInfo,
重采样性能度量中有缺失值。
我该怎么办?您的帮助对我意义重大。
我今天也遇到了同样的错误,后来我发现是因为我没有 library(randomForest)。希望这对其他遇到相同错误的人有所帮助。
感谢分享!
嗨,Jason,
当我运行 customRF 代码时
customRF$parameters <- data.frame(parameter = c("mtry", "ntree"), class = rep("numeric", 2), label = c("mtry", "ntree"))
customRF$grid <- function(x, y, len = NULL, search = "grid") {}
customRF$fit <- function(x, y, wts, param, lev, last, weights, classProbs, …) {
randomForest(x, y, mtry = param$mtry, ntree=param$ntree, …)
}
customRF$predict <- function(modelFit, newdata, preProc = NULL, submodels = NULL)
predict(modelFit, newdata)
customRF$prob <- function(modelFit, newdata, preProc = NULL, submodels = NULL)
predict(modelFit, newdata, type = "prob")
customRF$sort <- function(x) x[order(x[,1]),]
customRF$levels <- function(x) x$classes
# 训练模型
control <- trainControl(method="repeatedcv", number=10, repeats=3)
tunegrid <- expand.grid(.mtry=c(1:4), .ntree=c(100, 500, 1000))
set.seed(42)
custom <- train(SOH~., data=TrainSet, method=customRF, metric=metric, tuneGrid=tunegrid, trControl=control)
summary(custom)
plot(custom)
此时
custom <- train(SOH~., data=TrainSet, method=customRF, metric=metric, tuneGrid=tunegrid, trControl=control)
我收到此错误
Error: some required components are missing: library, type
这是什么意思?
另外,customRF 代码是否需要更改?或者我们可以直接复制粘贴吗?
抱歉,我以前没有见过这个错误。
您可以尝试在 stackoverflow 上搜索/发帖?
另一个值得尝试的包是“DEoptim”,它使用不同的进化算法进行“智能搜索”,这适用于浮点数和整数参数。
很好,谢谢分享!
当我运行
rf_default <- train(WOODLAND~., data=datasetgeo, method="rf", metric=metric, tuneGrid=tunegrid, trControl=control)
输出是
Error in
contrasts<-(*tmp*, value = contr.funs[1 + isOF[nn]])contrasts 只能应用于具有 2 个或更多级别的因子
您认为是什么问题?WOODLAND 的值是 0-125 的观测/计数。预测变量是各种环境变量,总共有 104 个。
很遗憾听到这个消息。我对那个数据集不熟悉,抱歉。
也许可以尝试在 cross validated 上发帖?
我运行的代码来自您提供的代码,用我的数据集运行但没有成功。x 数据集中的预测变量应该是都是因子还是都是数值,或者有区别吗?我能够运行您共享的所有代码行,直到 train 函数,输出显示正如我上面共享的那样,“contrasts 只能应用于具有 2 个或更多级别的因子”。我不确定“具有 2 个或更多级别的因子”是什么意思。y 数据/列值应该是因子,还是有区别?感谢您的时间和回复。
嗨 Jason
感谢发帖,非常有帮助。
在我成功调优参数(即找到最佳参数组合)后,如何对未见过的测试数据进行实际预测?
我是否需要将最佳参数作为输入用于 randomForest 函数
regr <- randomForest(x = X_train, y = y_train , mtry = 1, ntree = 10)
predictions <- predict(regr, X_test)
或者是否有办法直接使用新创建的 customRF 算法进行预测?
是的,通常使用 predict() 函数。
您好 Jason!非常感谢您精彩的文章!帮助很大!我正在尝试一种方法来调整 caret 中随机森林方法的最大树深度,但我没有在相关方法中看到任何相关的调优参数。唯一的调优参数是“mtry”。此外,我还使用了 for 循环来尝试不同的树值。是否有方法可以调整最大树深度?
我将非常感激您的善意合作。
也许您可以将参数作为网格搜索的一部分手动指定?
caret 文档可能包含更多信息
http://topepo.github.io/caret/index.html
从中学到了很多。我想问一下如何在 Extent caret 下为回归而不是分类创建一个新算法。我意识到讲座中的 customRF 是用于分类而不是回归,因为当我尝试时它给了我“Error: wrong model type for regression”。请问,您能好心发送 customRF 回归的代码,或者告诉我如何修改您提供的代码吗?谢谢。
嘿,Jason
我发现您的博客非常鼓舞人心!
并且 customRF 通过一些修改对回归有效。
我只有一个问题
为了对时间序列数据进行随机森林回归,在进行随机森林预测和调优参数之前,我们需要时间序列的平稳性,对吗?
我们是否还需要去除趋势和季节性?据我所知,随机森林可以在代码中处理这些问题?只有在 ARIMA 等情况下,我们才需要考虑这些因素?
谢谢
谢谢!
是的,在拟合模型之前使数据平稳是一个好主意。
是的,趋势和季节性平稳。
尝试制作和不制作系列平稳,并比较结果。
这是非常有帮助的内容。但是,对于大型输入,它需要很长时间,请在复制+粘贴之前小心!谢谢 Jason!
谢谢!
嗨,Jason,
在我的 PC 上 customRF 在多核上不起作用…您是否确认您遇到了同样的问题?
谢谢
Diego
听到这个消息我很难过。
也许暂时尝试单核运行?
集成…
它与
cores <- makeCluster(detectCores())
registerDoParallel(cores = cores)
但不是与
cl = makePSOCKcluster(detectCores())
registerDoParallel(cl)
在 centos 服务器上。
我不知道为什么。
Jason。为什么您在示例中使用 seed <- 7?您能进一步解释吗?
没有好理由,您可以使用任何您喜欢的数字。
各位
非常感谢您精彩的演示和讨论,这使其更有意义。
我正在寻找“在 R 中使用 NI(自然启发式)算法来调优 XGBoost”。
恕我直言,请分享。
致敬
Suraj
不客气。
抱歉,我无法提供帮助。
嗨,jason
当我在进行模型调优时
我看到了这个错误;
Error in data[, all.vars(Terms), drop = FALSE]
msgid “subscript out of bounds”
你能帮帮我吗?
此致
很遗憾听到这个消息。我以前从未见过这个错误。
也许可以尝试将您的代码和错误消息发布到 stackoverflow。
嗨,Jason,
我想知道您能否建议一种计算随机森林模型所需的最小样本量(或列数)的方法。您知道有什么经验法则吗?
先谢谢您了。
通常越多越好,这可能有帮助。
https://machinelearning.org.cn/much-training-data-required-machine-learning/
嗨,Jason,
不错的帖子!在您的自定义脚本中,您尝试了 ntree 的几个值进行调优:1000、1500、2000、2500。我想知道 ntreer 是否有理论范围?为什么您选择这些数字进行调优?我们如何理解这个超参数?更多的树是否更好?如果您能提供参考,那就太好了,提前感谢!
不,继续增加树的数量,直到性能停止改善。越多越好,但速度也越慢。
嗨,Jason,
感谢您的建议!在我的项目中,我尝试了 ntree = 500、1000、1500、2000 和 2500,但当我选择不同的 ntree 时,模型性能没有太大差异,但 mtry 似乎更重要。在这种情况下,您认为选择 ntree = 500 是安全的吗?再次感谢!
当然可以。
太棒了,谢谢 Jason!
不客气!
嗨,Jason,
感谢您提供这个非常有趣且具有教育意义的教程。我有两个问题,第一个是如何获得每次迭代的结果(数字)。
control <- trainControl(method="repeatedcv", number=10, repeats=3) or
control <- trainControl(method="LOOCV", number=10)
并在我们的主题中运行此模型
rf_default <- train(Class~., data=dataset, method="rf", metric=metric, tuneGrid=tunegrid, trControl=control)
如何获得每次运行的结果,即 rf_default 1…. rf_default 10
第二个是如何在网格搜索中添加权重
tunegrid <- expand.grid(.mtry=c(3, 4 ,5 ,6 , 7),
.ntree = c(500, 1000 ,1500,2000),
.nodesize = c(5,10,15),
.maxnodes = c(10,20,50),
.classwt = list(c(1,1),c(1,3),c(1,5)))
提前感谢你
Sidy
不客气。
好问题,我不知道。也许可以查看包的文档?
如何为网格搜索加权?这对我不合理,抱歉。
感谢您的回复。那么,在不平衡数据的情况下,在网格搜索最优参数时,我们试图给少数类更大的权重。
通常,模型在训练期间会被加权,而不是搜索过程。
老师您好,我看到了您在caret包中为randomforest算法调整树数量的代码。这里我有一个问题,您给的是分类问题的扩展代码。请您帮忙告诉我如何为回归类型的问题进行调整。
感谢您的建议,我将来可能会准备一个示例。
嗨,Jason,
您是否制作了回归的示例?
这将对我帮助很大😀
谢谢
你好Chloe…我强烈推荐以下资源来帮助您解决可能遇到的许多问题。
https://machinelearning.org.cn/r-bundle/
嗨,Jason,
关于机器学习的很棒的教程。我想知道是否有可能绘制ROC曲线
在上述iris数据集的n折交叉验证测试上。
任何帮助将不胜感激。
谢谢你
谢谢!
不,通常ROC曲线是为单个测试集绘制的。
Jason及各位好!
很棒的教程!我从调优随机森林中学到了很多。
我想知道是否有原因导致调优mtry和ntree对我们模型的准确性影响最大?
任何帮助将不胜感激。
祝好!
它们是影响该算法最大的两个超参数。
你好,您是否有涵盖Caret ranger和Random Forest Rule-Based Model实现随机森林的调整参数的脚本?
我不确定,抱歉,也许可以尝试在博客上搜索或者改编上面的例子。
你好,Jason。
感谢教程,但我难以重现您得到的确切值。此示例是在哪个版本的R和caret下运行的?
感谢您制作这个。
版本应该不重要。由于以下原因,您可能永远无法得到与所示相同的值:https://machinelearning.org.cn/different-results-each-time-in-machine-learning/
mtry函数到底做什么?您能举个简单的例子吗。
“mtry:每次拆分时随机采样的变量数量。”
这是随机森林算法中的一个超参数。
很好的文章,易于理解。您在谈论树的数量和ntree值之间的交互作用。您具体指的是什么?它可能是例如ntree和RMSE(或MSE、R2等)吗?有没有办法在图表中可视化这些交互作用?
你好Nikolas…是的,交互作用可以用RMSE来衡量。这是首选方法。
很有帮助,但自定义调优的解释很差。没有解释“wts”、“lev”、“last”等是什么意思以及如何使用它们。我想将类别或案例权重纳入其中,但一直无法弄清楚。
Trenton,感谢您的反馈!